Des chercheurs de l’Institut Pasteur et du CNRS, en collaboration avec une équipe CNRS/Inserm/Université Paris Descartes-Sorbonne Paris Cité (située à l’Institut Cochin), ainsi qu’avec le Wellcome Trust (Sanger Institute), viennent de découvrir la cause de l’émergence des infections néonatales à streptocoque B dans les années 1960. Ces résultats, publiés dans la revue Nature Communications, le 4 août, apportent la preuve que l'apparition soudaine de ces infections provoquées par cette bactérie est due à l’usage massif, à partir des années 1950, d’un antibiotique, la tétracycline.
Communiqué de presse
Paris, le 4 août 2014
Le streptocoque du groupe B (Streptococcus agalactiae) est à l’origine de graves infections néonatales, concernant environ une naissance sur 2 000. Il a pour réservoir principal le tube digestif à partir duquel se fait la colonisation des voies urogénitales. Lors de l'accouchement, la mère peut transmettre la bactérie pathogène au nouveau-né. La prévention de ces infections repose sur un dépistage en fin de grossesse du portage de la bactérie. Si le test est positif, un traitement préventif par antibiotique est administré à la mère pendant l’accouchement.
Dans les années 1960, aux États-Unis et en Europe, une augmentation des infections néonatales dues à streptocoque B a été rapportée dans les registres des maternités. L'absence d'infections au streptocoque B reportées jusqu'alors pouvait résulter d'un sous-diagnostic ou de sous-déclaration de ces infections. Néanmoins, s'il y avait émergence, ses causes étaient inconnues.
Les chercheurs de l'équipe Institut Pasteur/CNRS dirigée par Philippe Glaser en collaboration avec une équipe CNRS/Inserm/Université Paris Descartes-Sorbonne Paris Cité (située à l’Institut Cochin), des chercheurs de l’Assistance Publique - Hôpitaux de Paris (AP-HP), du Wellcome Trust (Sanger Institute) et des microbiologistes d'Europe et d'Australie, sont parvenus à répondre à cette énigme en retraçant l’histoire évolutive des streptocoques B d'origine humaine. Pour cela, ils ont séquencé et comparé le génome de 230 souches datant des années 1950 à aujourd’hui, et établi leurs relations évolutives. Ils ont constaté que la population des streptocoques B colonisant et infectant l'homme est constituée d'un petit nombre de clones présentant une très faible diversité génétique. Cette caractéristique témoigne d’une origine commune récente en accord avec un phénomène d'émergence.
Par ailleurs, de manière remarquable, 90 % des souches de streptocoques B isolées chez l'homme sont résistantes à la tétracycline. L'analyse génomique des clones a permis aux chercheurs d'identifier le support génétique de cette résistance et surtout de montrer que ces clones, identifiés par l'étude évolutive, avaient été sélectionnés pour leur résistance à cet antibiotique. En accord avec cette observation, ils ont pu dater leur émergence au milieu du 20ème siècle. Cette époque fait suite à l’usage massif, dans les années 1950, d'un des premiers antibiotiques, administré à titre préventif et curatif contre diverses infections : la tétracycline. Sur la base de ces résultats, les chercheurs suggèrent que l’utilisation de cet antibiotique a entrainé le remplacement global d'une population sensible de streptocoques B, peu dangereux, par quelques clones résistants qui se sont disséminés sur les cinq continents. Ces clones, qui auraient été sélectionnés également pour leurs propriétés de dissémination et de colonisation, sont actuellement responsables des infections observées à partir des années 1960.
Ces résultats élucident les raisons de l'émergence des infections humaines à streptocoque B et démontrent l'impact délétère à long terme de l'utilisation massive et non contrôlée des antibiotiques. En effet, la tétracycline, qui n'a jamais été utilisée pour le traitement des infections à streptocoque B et n’est plus depuis 20 ans que rarement prescrite chez l'homme, a eu un impact irréversible sur cette population bactérienne qui reste une des premières causes d'infections néonatales. A l’heure où la multi-résistance des bactéries est un problème crucial, cette étude souligne la nécessité de prendre en compte pour tout antibiotique prescrit, non seulement le risque direct du développement de la multi-résistance, mais aussi son impact sur les populations bactériennes constituant les flores normales de l'homme et des animaux.
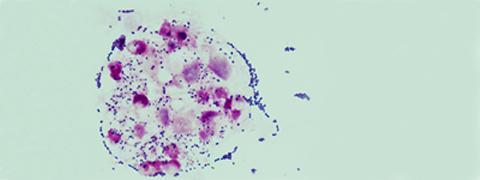
Illustration : Streptococcus agalactiae dans des globules blancs (polynucléaires neutrophiles) dans un liquide céphalorachidien d’un enfant atteint de méningite (coloration de Gram microscopie optique). © Claire Poyart, CNR-Strep (AP-HP, Inserm)
Source
Streptococcus agalactiae clones infecting humans were selected and fixed through the extensive use of tetracycline, Nature Communications, 1er août 2014, DOI: http://dx.doi.org/ 10.1038/ncomms5544
(1,2,3‡) Violette Da Cunha, (4,5,‡) Mark R. Davies, (1,2) Pierre-Emmanuel Douarre, (1,2) Isabelle Rosinski-Chupin, (6) Immaculada Margarit, (7) Sebastien Spinali, (6) Tim Perkins, (3) Pierre Lechat, (7) Nicolas Dmytruk, (1,2) Elisabeth Sauvage, (8) Laurence Ma, (6) Benedetta Romi, (8) Magali Tichit, (1,2) Maria-José Lopez-Sanchez, (3) Stéphane Descorps-Declere, (3) Erika Souche, (2,9) Carmen Buchrieser, (1,10) Patrick Trieu-Cuot, (3) Ivan Moszer, (11) Dominique Clermont, (6) Domenico Maione, (8) Christiane Bouchier, (12, 13) David J. McMillan, (4) Julian Parkhill, (6) John L. Telford, (4) Gordan Dougan, (5) Mark J. Walker, #The DEVANI Consortium, (4) Matthew T. G. Holden, (1, 7,14,15) Claire Poyart and (1,2,3 *) Philippe Glaser
(1) Institut Pasteur, Unité de Biologie des Bactéries Pathogènes à Gram-positif, Paris, France;
(2) CNRS UMR3525, Paris, France;
(3) Institut Pasteur, Bioinformatics platform, Paris, France;
(4) The Wellcome Trust Sanger Institute, Hinxton, Cambridge, UK;
(5) Australian Infectious Diseases Research Centre, School of Chemistry and Molecular Biosciences, University of Queensland, Australia;
(6) Novartis Vaccines and Diagnostics, Siena, Italy;
(7) Centre National de Référence des Streptocoques, Groupe Hospitalier Paris Centre Cochin - Hôtel Dieu-Broca, Paris, France;
(8) Institut Pasteur Genomic platform, Paris, France;
(9) Institut Pasteur, Biologie des Bactéries Intracellulaires, Paris, France;
(10) CNRS ERL3526, Paris, France;
(11) Institut Pasteur, Collection de l'Institut Pasteur (CIP), Paris, France;
(12) QIMR Berghofer Medical Research Institute, Brisbane, Australia;
(13) Inflammation and Healing Research Cluster, University of the Sunshine Coast, Sippy Downs, Australia;
(14) Institut Cochin, Université Sorbonne Paris Descartes;
(15) INSERM, U1016, Paris, France.
‡These authors contributed equally to this work