Des chercheurs de l’Institut Pasteur, de l’INRA, de l’Inserm et du CNRS viennent d’identifier un mécanisme qui permet à la bactérie pathogène Listeria monocytogenes de reprogrammer à son avantage l’expression des gènes de la cellule qu’elle infecte. L. monocytogenes sécrète une protéine capable de pénétrer dans le noyau des cellules afin de prendre le contrôle de gènes du système immunitaire de l’hôte. Ces travaux ont été publiés sur le site de la revue Science le 20 janvier 2011.
Communiqué de presse
Paris, le 25 janvier 2011

Ces travaux s’inscrivent dans la lignée d’une étude réalisée par la même équipe en 2009. Celle-ci avait permis l’identification d’un complexe capable de verrouiller l’expression des gènes en compactant l’ADN (2). Ici, les chercheurs ont identifié une petite protéine bactérienne, nommée LntA, capable de faire sauter ce verrou en se fixant directement sur le complexe, ce qui provoque l’ouverture de l’ADN compacté et donc l’accès aux gènes.
On ignore encore comment, et à quel moment, la bactérie décide de la production de ce facteur LntA, mais son expression est indispensable au bon déroulement de l’infection par Listeria, qui peut grâce à elle activer ou réprimer à sa guise l’immunité de l’hôte.
Ces travaux laissent entrevoir le rôle d’une régulation épigénétique - des changements dans l’expression des gènes, ayant lieu sans altération de la séquence ADN - dans l’infection par L. monocytogenes. Cette découverte, si elle se vérifiait pour d’autres pathogènes, apporterait de précieuses informations permettant de mieux comprendre et, à terme, de mieux lutter, contre les maladies infectieuses et immunitaires.
Cette étude a reçu notamment le soutien financier de la Communauté européenne (programmes ERANET PathoGenomics et ERC).
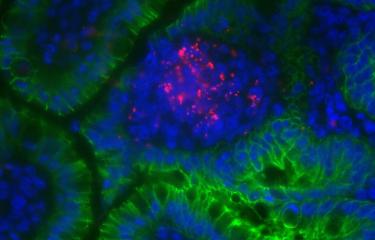
Figure : Protéine bactérienne LntA (en vert-jaune) localisée dans le noyau de deux cellules humaines infectées par Listeria (en violet-bleu).
Source
(1) A Bacterial Protein Targets the BAHD1 Chromatin Complex to Stimulate Type III Interferon Response, Science en ligne, 20 janvier 2011
Alice Lebreton (1,2,3), Goran Lakisic (4), Viviana Job (5), Lauriane Fritsch (6), To Nam Tham (1,2,3), Ana Camejo (7), Pierre-Jean Matteï (5), Béatrice Regnault (8), Marie-Anne Nahori (1,2,3), Didier Cabanes (7), Alexis Gautreau (4), Slimane Ait-Si-Ali (6), Andréa Dessen (5), Pascale Cossart (1,2,3) and Hélène Bierne (1,2, 3)
(1) Institut Pasteur, Unité des Interactions Bactéries Cellules, Paris, F-75015 France;
(2) Inserm, U604, Paris, F-75015 France;
(3) INRA, USC2020, Paris, F-75015 France.
(4) CNRS UPR3082, Laboratoire d’Enzymologie et de Biochimie Structurales, Gif-sur- Yvette, F-91198 France.
(5) Institut de Biologie Structurale, Bacterial Pathogenesis Group, UMR 5075 (CNRS/CEA/UJF), Grenoble, France.
(6) CNRS UMR7216, Université Paris-Diderot / Paris 7, Paris, F-75013 France.
(7) Institute for Molecular and Cell Biology, Porto, Portugal.
(8) Institut Pasteur, Génopole, Paris, F-75015 France.
(2) « Human BAHD1 promotes heterochromatic gene silencing », PNAS, 2009, vol. 106 _ no. 33 pp. 13826-13831.
Hélène Bierne, To Nam Tham, Eric Batsche, Anne Dumay, Morwenna Leguillou, Sophie Kernéis-Golsteyn, Béatrice Regnault, Jacob-S Seeler, Christian Muchardt, Jean Feunteun and Pascale Cossart.
Contact presse
Service de presse de l’Institut Pasteur
Sabine D'Andrea - 01 44 38 92 17 - sabine.dandrea@pasteur.fr
Nadine Peyrolo - 01 45 68 81 47 - nadine.peyrolo@pasteur.fr