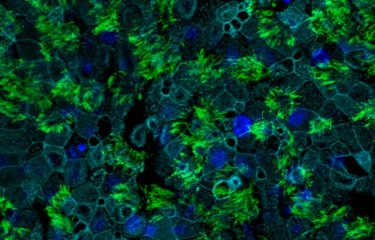
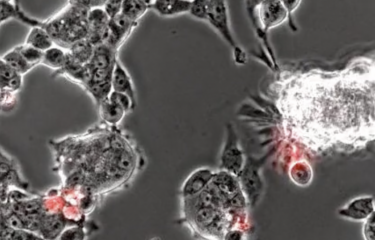
Des scientifiques ont mis à jour les endroits précis des cellules respiratoires où se fixe le virus HKU1, permettant ainsi d’expliquer en détail le mécanisme d’infection mis en œuvre. Une découverte qui a permis d’en savoir plus sur les éventuelles autres espèces animales abritant ce virus mais également de développer de premiers anticorps bloquant l’infection.
Par quels mécanismes exacts les coronavirus nous infectent-ils ? Plus de quatre ans après l’émergence chez l’être humain du tristement célèbre SARS-CoV-2, de nombreux scientifiques continuent de plancher sur la question. À ce jour, il est communément admis que nous pouvons être infectés par sept coronavirus différents. Certains peuvent être à l’origine de pneumopathies ou d’effets graves sur la santé, comme le SARS-CoV-2 -à l’origine de la Covid-19, mais également le MERS-CoV ou le SARS-CoV-1. D’autres, comme le virus HKU1, provoquent des maladies plus bénignes comme des rhumes courants.
La structure moléculaire pour mieux comprendre l’infection par HKU1
Concernant ce dernier virus, si une étape importante avait été franchie il y a quelques mois avec l’identification de TMPRSS2 comme molécule réceptrice permettant à HKU1 de se fixer sur nos cellules, les mécanismes précis de l’infection restaient à découvrir. Dans une étude coordonnée par des scientifiques de l’Institut Pasteur, les différentes étapes permettant l’entrée de HKU1 dans nos cellules ont été dévoilées. Pour cela, “nous avons obtenu une structure en 3D, à l’échelle de l’atome, du complexe formé entre TMPRSS2 et la partie de la spicule de HKU1 se liant au récepteur. Nous avons ainsi pu décrire les zones de contact des deux protéines et les changements de conformation de celles-ci qui permettent l’entrée du virus dans la cellule cible” explique Félix Rey, responsable de l’unité Virologie structurale et co-dernier auteur de l’étude. “Nous avons ainsi pu mettre en évidence que les mécanismes d’entrée dans la cellule sont différents pour HKU1 et pour les autres coronavirus, tels que le SARS-CoV-2.”
Structure tridimensionnelle du complexe moléculaire entre le récepteur cellulaire TMPRSS2 (en rose) et le domaine de liaison au récepteur de la protéine de spicule du coronavirus HKU1 (en bleu) © Institut Pasteur/Ignacio Fernandez
Les espèces animales porteuses du virus restent à découvrir
Si la connaissance précise de la structure du complexe moléculaire permet de mieux comprendre le processus d’infection, elle apporte également d’autres informations. Elle permet d’identifier les parties critiques du récepteur cellulaire TMPRSS2... et de voir si elles sont également présentes chez d’autres espèces animales. Les scientifiques ont comparé les séquences des équivalents de TMPRSS2 chez plus de 200 espèces de mammifères. Ils ont ainsi pu identifier, à deux positions précises du récepteur cellulaire humain, deux acides aminés indispensables pour l’infection par HKU1 et qui ne sont pas présents chez les autres animaux. Selon Olivier Schwartz, responsable de l’unité Virus et immunité et co-dernier auteur de l’étude, “le macaque, le cochon, la souris, le furet ou encore le hamster, entre autres, ne sont donc probablement pas des réservoirs animaux pour ce virus”. En d’autres termes, en ce qui concerne l’origine animale de HKU1, l’enquête est toujours en cours.
Des anticorps pour empêcher l’infection
Si, sur la question de l’origine du virus les investigations doivent se poursuivre, ces travaux ont également ouvert la voie à des outils thérapeutiques, préventifs ou diagnostiques. Les scientifiques ont notamment réussi à produire un mini-anticorps –ou nanobody (nAb)- anti-TMPRSS2. La structure du complexe nAb/TMPRSS2 révèle que cet anticorps se fixe sur le récepteur cellulaire au même endroit que la spicule de HKU1. Le nanobody, “en prenant la place” de HKU1 sur le récepteur cellulaire, empêche la fixation du virus sur la cellule et l’infection se retrouve bloquée. Une première piste préventive à développer. Enfin, si TMPRSS2 est le point d’ancrage cellulaire de HKU1, c’est également une enzyme retrouvée dans plusieurs processus physiologiques. Ces mini-anticorps anti-TMPRSS2 pourraient donc représenter à terme un outil thérapeutique ou d’aide au diagnostic de choix dans le cadre de diverses pathologies.
Ces travaux ont été lancés et financés par l’Institut Pasteur début 2020 en réponse à la pandémie de Covid-19. Plusieurs projets de recherche en lien avec le SARS-CoV-2 et d’autres coronavirus sont toujours en cours.
Structural basis of TMPRSS2 zymogen activation and recognition by the HKU1 seasonal coronavirus, Cell, 3 juillet 2024
Ignacio Fernández1,8, Nell Saunders2,8, Stéphane Duquerroy1,3, William H. Bolland2, Atousa Arbabian1, Eduard Baquero4, Catherine Blanc5, Pierre Lafaye6, Ahmed Haouz7, Julian Buchrieser2, Olivier Schwartz2,*, Félix A. Rey1,9,*
1 Institut Pasteur, Université de Paris Cité, CNRS UMR 3569, Structural Virology Unit, 75015, Paris, France.
2 Institut Pasteur, Université de Paris Cité, CNRS UMR 3569, Virus & Immunity Unit, 75015, Paris, France and Vaccine Research Institute, Créteil, France
3 Université Paris-Saclay, Faculté des Sciences, Orsay, France.
4 Institut Pasteur, Université de Paris Cité, INSERM U1222, Nanoimaging core, 75015, Paris, France.
5 Institut Pasteur, Université de Paris Cité, Pasteur-TheraVectys Joint Lab, Paris, France
6 Institut Pasteur, Université Paris Cité, CNRS UMR 3528, Antibody Engineering Facility-C2RT, 75015, Paris, France.
7 Institut Pasteur, Université Paris Cité, CNRS UMR 3528, Crystalogenesis Facility-C2RT, 75015, Paris, France.
8Equal contribution
9Lead contact