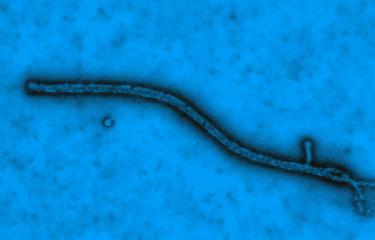
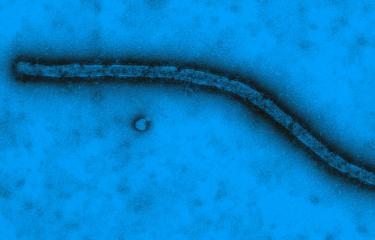
Dans un article paru le 16 avril dans The New England journal of Medicine, des chercheurs de l'Inserm (Laboratoire P4 Jean Mérieux-Inserm de Lyon) et de l'Institut Pasteur publient leurs premières conclusions sur les caractéristiques du virus Ebola découvert en Guinée. Les premières investigations virologiques ont permis d'identifier le Virus Ebola Zaïre comme étant l'agent pathogène responsable de cette épidémie. Menés en moins d'un mois, le séquençage entier du génome et son analyse phylogénétique montrent que le virus présent en Guinée forme un clade (variant) distinct des souches identifiées par le passé en République démocratique du Congo et au Gabon. Leurs investigations épidémiologiques ont également fait le lien entre les cas confirmés en laboratoire et les premiers décès recensés lors de l'épisode de décembre 2013.
Le Virus Ebola est un virus mortel très contagieux pour lequel il n'existe aucun traitement à ce jour. Les symptômes sont peu spécifiques puisqu'il s'agit de fièvre, de diarrhée sévère et de vomissements. 30 à 90 % des personnes infectées meurent des suites de cette infection.
Le 2 avril 2014, l’Organisation mondiale de la santé (OMS) indiquait, dans un communiqué publié par l’agence de l’ONU, avoir dénombré 5 nouveaux cas de fièvre Ebola en Guinée. Le bilan de cas suspects et confirmés de la flambée de fièvre Ebola en cours depuis janvier en Guinée est de 127 cas, dont 83 décès, selon l’OMS qui précise que 35 cas ont été confirmés par des tests de laboratoire.
Les premiers échantillons ont été analysés à Lyon dans le laboratoire P4 Jean Mérieux-Inserm dirigé par Hervé Raoul, directeur de recherche Inserm, par le Centre national de référence des fièvres hémorragiques virales (rattaché à l’unité de biologie des infections virales émergentes de l’Institut Pasteur, dirigée par Sylvain Baize). Un diagnostic positif a été établi.
Un laboratoire P4 mobile a été déployé en Guinée pour apporter sur le terrain une aide à la réalisation du diagnostic. Ce laboratoire mobile a été développé dans le cadre d’un projet européen “EMLab″ coordonné par des chercheurs allemands et dont le laboratoire P4 Jean Mérieux-Inserm est le partenaire français.
Des échantillons sanguins prélevés chez 20 patients ont pu être analysés par les chercheurs. Divers tests ont été menés par les scientifiques afin d'établir une carte d'identité spécifique du virus.
L'ARN viral a été extrait des échantillons sanguins, amplifié puis séquencé. Ces séquences ont enfin été comparées à 48 génomes entiers déjà connus de virus Ebola. D'après les résultats, l'analyse a montré 97 % de similitudes avec les souches identifiées en république démocratique du Congo en 1976 et 2007 et au Gabon en 1994 et 1996.
"Ces résultats démontrent que nous sommes face à l'émergence d'une nouvelle "forme" de ce virus en Guinée" explique Hervé Raoul directeur du laboratoire P4. Celle-ci est commune aux cas découverts depuis le mois de décembre.
Il semblerait qu’une seule introduction de l’animal à l’homme soit à l’origine de cette épidémie.
Au-delà de l’épidémie en cours, ces résultats démontrent que la zone de circulation du virus Ebola est plus large que ce qui était connu auparavant, avec comme conséquence que l’Afrique de l’Ouest doit être dorénavant considérée comme une zone à risque pour le virus Ebola. Des mesures visant à prévenir la transmission de la faune sauvage à l’homme et à identifier rapidement de tels évènements s’ils venaient à se reproduire devront être mis en place en Guinée, mais aussi dans les pays limitrophes.
Pour en savoir plus sur le laboratoire P4
Le laboratoire P4 Jean Mérieux est un laboratoire de haut confinement dédié à l’étude des agents pathogène de classe 4. Le niveau de sécurité biologique qui y est appliqué est de 4, niveau le plus élevé. Les chercheurs y travaillent équipés d’un scaphandre maintenu en surpression pour les protéger de toute contamination. Le laboratoire est lui-même maintenu en dépression afin de protéger l’environnement. De plus, tous les déchets produits sont totalement inactivés et l’air extrait est purifié par un système de double filtration absolue. Ce laboratoire est encore aujourd’hui la structure de ce niveau de confinement offrant la plus grande capacité d’expérimentation en Europe.
Des agents hautement pathogènes
Les agents pathogènes de classe 4 (ou de groupe de risque 4) sont des microorganismes hautement pathogènes caractérisés par un taux de mortalité très élevé, l’absence d’outils prophylactiques ou thérapeutiques pour s’en protéger et leur facilité de transmission. Les agents pathogènes de classe 4 connus à ce jour sont tous des virus responsables notamment de fièvres hémorragiques ou d’encéphalites. On compte parmi eux les virus Ebola, Marburg, Lassa, Junin, Machupo, Guanarito, Sabia, Crimée-Congo, Nipah et Hendra.
Pour obtenir des photos du laboratoire P4 : http://www.serimedis.inserm.fr/fr/feature/content/id/989
Sources
Emergence of Zaire Ebola Virus Disease in Guinea — Preliminary Report
Sylvain Baize, Ph.D., Delphine Pannetier, Ph.D., Lisa Oestereich, M.Sc.,Toni Rieger, Ph.D., Lamine Koivogui, Ph.D., N’Faly Magassouba, Ph.D., Barrè Soropogui, M.Sc., Mamadou Saliou Sow, M.D., Sakoba Keïta, M.D., Hilde De Clerck, M.D., Amanda Tiffany, M.P.H., Gemma Dominguez, B.Sc.,
Mathieu Loua, M.D., Alexis Traoré, M.D., Moussa Kolié, M.D., Emmanuel Roland Malano, M.D., Emmanuel Heleze, M.D., Anne Bocquin, M.Sc., Stephane Mély, M.Sc., Hervé Raoul, Ph.D., Valérie Caro, Ph.D., Dániel Cadar, D.V.M., Ph.D., Martin Gabriel, M.D., Meike Pahlmann, Ph.D., Dennis Tappe, M.D., Jonas Schmidt-Chanasit, M.D., Benido Impouma, M.D., Abdoul Karim Diallo, M.D., Pierre Formenty, D.V.M., M.P.H., Michel Van Herp, M.D., M.P.H., and Stephan Günther, M.D.
New England Journal of Medicine, 16 avril 2014, http://dx.doi.org/10.1056/NEJMoa1404505
Contact chercheur
Inserm
Delphine Pannetier
04 72 76 82 91
delphine.pannetier@inserm.fr
Institut Pasteur
Sylvain Baize
04 37 28 24 43,
sylvain.baize@pasteur.fr
Contact presse