L’équipe de gastroentérologie de l’hôpital Henri-Mondor AP-HP et de l’Université Paris-Est Créteil, dirigée par le Pr Iradj Sobhani, et l’équipe de l’Inserm et de l’Institut Pasteur U1202 « Unité de pathogénie microbienne moléculaire », dirigées par le Pr Philippe Sansonetti, qui occupe également la chaire Microbiologie et maladies infectieuses du Collège de France, ont montré qu’un déséquilibre du microbiote intestinal, appelé « dysbiose », favorisait la survenue d’un cancer du côlon. Les équipes françaises, sont réunies sous le label Oncomix depuis avril 2016 ; ces équipes en collaboration avec l’équipe d’immunologie des cancers de Mayo Clinic dirigé par le Pr Khazaie aux Etats-Unis ont en effet montré que la transplantation de flore fécale de patients atteints d’un cancer colique chez la souris causait des lésions et des modifications épigénétiques caractéristiques du développement d’une tumeur maligne.
L’étude pilote, financée par l’Institut national du cancer et promue par l’AP-HP dans le cadre d’un programme hospitalier de recherche clinique en cancérologie (PHRC-K), a ainsi permis de concevoir un test sanguin non invasif qui identifie ce phénomène épigénétique associé à la dysbiose et de le valider chez 1000 personnes. Les résultats ont fait l’objet le 11 novembre 2019 d’une publication dans la revue Proceedings of the National Academy of Sciences (PNAS).
Le cancer colorectal sporadique (CCR) se développe sans que les facteurs de risques puissent être déterminés. Il résulte d’interactions complexes entre le sujet et son environnement. L’augmentation croissante de son incidence suggère une évolution défavorable de l’environnement, dont les composantes peuvent induire des altérations d’ADN (génique et épigénique) des cellules de l’hôte pour les cancers colorectaux sporadiques.
Plusieurs études se sont intéressées au rôle du microbiote en tant que médiateur de ces interactions. L’équipe du service de gastroentérologie de l’hôpital Henri-Mondor AP-HP et de l’Université Paris-Est Créteil, et une équipe de l’Inserm et de l’Institut Pasteur U1202 « Unité de pathogénie microbienne moléculaire » et de la chaire de microbiologie et maladies infectieuses du Collège de France, ont démontré chez la souris, puis validé chez l’homme, que le mécanisme épigénétique favorisé par certaines bactéries du microbiote contribuait à la survenue ou la promotion des cancers colorectaux sporadiques.
Les 136 souris inclues dans l’étude recevaient par transplantation soit des selles fraiches de neuf patients atteints de cancers colorectaux sporadiques, soit des selles fraiches de neuf patients sans anomalie colique pris en charge à l’hôpital Henri-Mondor AP-HP. Leurs colons ont été examinés 7 et 14 semaines après la transplantation de microbiote fécal (TMF) humain. Les équipes se sont notamment intéressées au nombre et à l’évolution des lésions précancéreuses-cryptes aberrantes (CA), au profil microbien et aux altérations d’ADN colique. Ils ont aussi pris en compte la prise alimentaire, le poids et les indicateurs biologiques sanguins des animaux.
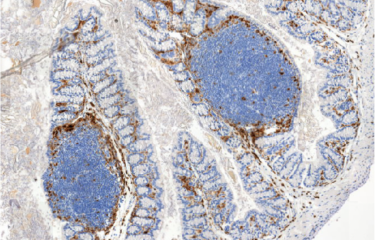
Des associations entre la signature d'ADN (génique et épigénique) tissulaire et la dysbiose fécale (qui correspond à un déséquilibre dans la composition bactérienne intestinale) ont été identifiées par des tests statistiques. Les souris, qui avaient reçu des selles fraiches de patients atteints de cancers colorectaux sporadiques, ont développé des lésions précancéreuses, dites « cryptes aberrantes » (CA) sans modification génétique significative colique mais elles présentaient un plus grand nombre de gènes hyperméthylés. Ces derniers ont été significativement liés à la survenue de CA au sein de la muqueuse colique.
Après vérification des associations entre dysbiose fécale, et anomalies d’ADN (méthylation) chez les patients atteints d’un cancer colorectal sporadique et ayant participé au transfert fécal, une étude pilote a été menée chez l'homme afin de mettre au point un test sanguin simple et reproductible permettant de diagnostiquer les tumeurs colorectales au stade précoce chez des patients asymptomatiques. Sa validation prospective a été réalisée chez 1000 patients asymptomatiques qui devaient bénéficier d’une coloscopie. Pour identifier les bactéries impliquées, l’ensemble de leur génome bactérien a été séquencé. Le niveau d’hyperméthylation de trois gènes a été défini comme un index cumulé de méthylation (CMI). Les patients ont été classés en fonction de leur CMI (positif ou négatif). Une analyse a identifié le CMI positif comme un facteur prédictif de la survenue d’un cancer colorectal sporadique.
Ces travaux montrent ainsi que le microbiote des sujets présentant un cancer colorectal sporadique induit des lésions précancéreuses coliques chez l’animal par l’hyperméthylation d’un nombre restreint de gènes. Le taux cumulé d’hyperméthylation (CMI) et/ou les bactéries pro méthylatrices pourraient ainsi devenir des marqueurs diagnostiques de ce type de cancer. Ces premiers résultats devront être évalués et confirmés dans le cadre d’un essai clinique.
Source
Colorectal cancer-associated microbiota contributes to oncogenic epigenetic signatures, PNAS, 11 novembre 2019
Iradj Sobhania,b,1, Emma Bergstena,c, Séverine Couffina, Aurélien Amiota,b, Biba Nebbadd, Caroline Baraue,Nicola de’Angelisf, Sylvie Rabotg, Florence Canoui-Poitrineh, Denis Mestiviera,i, Thierry Pédronc, Khashayarsha Khazaiej,2, and Philippe J. Sansonettic,k,1,2
a EA7375 (EC2M3 Research Team), Université Paris Est, Créteil 94000, France;
b Service de Gastroenterologie, Hôpital Henri Mondor, Assistance Publique-Hôpitaux de Paris, Créteil 94000, France;
c Unité de Pathogénie Microbienne Moléculaire, INSERM U1202, Institut Pasteur, Paris 75015, France;
d Service deMicrobiologie, Hôpital Henri Mondor, Assistance Publique-Hôpitaux de Paris, Créteil 94000, France;
e Plateforme de Ressources Biologique, Hôpital HenriMondor, Créteil 94000, France;
f Service de Chirurgie Digestive, Hôpital Henri Mondor, Assistance Publique-Hôpitaux de Paris, Créteil 94000, France;
g MicalisInstitute, Institut National de la Recherche Agronomique (INRA), AgroParisTech, Université Paris-Saclay, Jouy-en-Josas 78352, France;
h Service de SantéPublique, Hôpital Henri Mondor, Assistance Publique-Hôpitaux de Paris, Créteil 94000, France;
i Bioinformatics Core Lab, INSERM U955, Institut Mondor deRecherche Biomédicale, Créteil 94000, France;
j Department of Immunology, Mayo Clinic, Rochester, MN 55905; and
k Chaire de Microbiologie et Maladies Infectieuses, Collège de France, Paris 75005, France