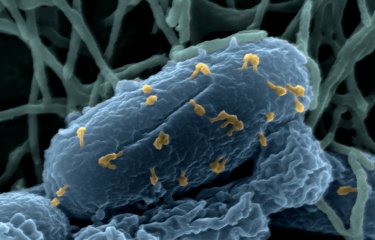
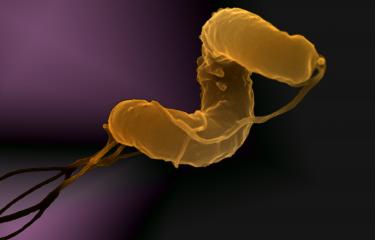
Avec l’essor des antibiotiques dans les années 1930, la phagothérapie (c’est à dire l’utilisation de virus appelés bactériophages dans la lutte contre les infections bactériennes) a été abandonnée. Aujourd’hui, la montée de l’antibiorésistance rend le traitement des infections bactériennes de plus en plus difficile et la phagothérapie suscite à nouveau l’intérêt des médecins et des chercheurs malgré sa complexité de mise en œuvre due à la très grande diversité et spécificité des bactériophages. C’est ainsi que des scientifiques de l’Institut Pasteur, de l’Inserm, de l’AP-HP et de l’Université Paris Cité ont développé un nouvel outil susceptible de choisir, de façon simple et efficace, le meilleur cocktail de bactériophages possible pour un patient donné. Pour cela, ils ont élaboré et entraîné un modèle à base d’intelligence artificielle capable de sélectionner sur mesure des bactériophages en se basant uniquement sur le génome des bactéries ciblées. Les résultats de ces travaux ont été publiés le 31 octobre 2024 dans la revue Nature Microbiology, et ouvrent la voie à des phagothérapies personnalisées pour combattre des infections bactériennes résistantes aux antibiotiques.
Certaines bactéries, comme Escherichia coli, se montrent de plus en plus résistantes aux antibiotiques classiques et deviennent ce que l’on appelle des « superbactéries ». Pour contourner ces résistances, qui représentent un problème de santé publique majeur, des équipes de recherche explorent la voie de la phagothérapie. Le principe : utiliser des virus, appelés phages ou bactériophages, qui n’infectent que des bactéries pour éliminer de façon ciblée celles pathogènes pour l’homme. « La phagothérapie a été inventée par le chercheur pasteurien Félix d'Hérelle dans les années 1920 puis a été abandonnée avec l’essor des antibiotiques à la fin des années 1930, beaucoup plus simples et économiques à fabriquer et à utiliser. Aujourd’hui, seuls quelques pays de l’Europe de l’Est, comme la Géorgie, utilisent encore la phagothérapie, tandis que dans les pays occidentaux, des phages à "large spectre" sont utilisés ponctuellement de façon compassionnelle pour traiter des infections chroniques multirésistantes aux antibiotiques(1), quand plus aucun médicament autorisé n’est efficace, rappelle Baptiste Gaborieau, co-premier auteur de l’article, médecin réanimateur à l’Hôpital Louis Mourier (AP-HP) et chercheur dans le laboratoire IAME (Université Paris Cité-Inserm). Depuis une vingtaine d’années, grâce à sa promotion par l’OMS(2) et plus récemment la mise en place d’essais cliniques notamment européens, la phagothérapie suscite à nouveau l’intérêt. »
L’un des défis est de savoir quel bactériophage sera efficace pour lutter contre une infection donnée, sachant que chaque phage ne peut infecter que certaines souches(3) bactériennes. Dans le sol ou l’eau où les phages sont présents naturellement, ils circulent jusqu’à trouver la bonne cible. C’est ainsi que des scientifiques de l’Institut Pasteur, de l’Inserm, de l’AP-HP et de l’Université Paris-Cité, ont décidé d’étudier de plus près les interactions bactéries-phages afin de savoir s’il était possible de prédire l’efficacité d’un bactériophage sur une souche bactérienne donnée. La première étape a donc consisté en la création d’une base de données de qualité avec d’un côté 403 souches de bactéries Escherichia coli et de l’autre 96 bactériophages. Un travail qui aura nécessité plus de deux ans d’efforts. « Nous avons mis en contact les phages avec les bactéries en culture et observé quelles bactéries étaient tuées. Nous avons étudié 350 000 interactions et réussi à identifier, au niveau du génome des bactéries, les caractéristiques susceptibles de prédire l’efficacité des phages, résume Aude Bernheim, principale autrice de l’étude et responsable du laboratoire Diversité moléculaire des microbes à l’Institut Pasteur. « Contrairement à ce que l’on pensait initialement, ce sont les récepteurs à la surface des bactéries et non leurs mécanismes de défenses qui déterminent en premier lieu la capacité des bactériophages à pouvoir ou non infecter les bactéries, et qui présagent de leur efficacité », poursuit Florian Tesson, co-premier auteur de l’article et doctorant dans les laboratoires Diversité moléculaire des microbes à l’Institut Pasteur et IAME à l’Université Paris Cité-Inserm.
Grâce à cette analyse précise et complète des mécanismes d’interaction entre les bactéries et les phages, les bio-informaticiens de l’équipe ont pu concevoir un programme d’intelligence artificielle optimisé et efficace. Ce dernier se base sur l’analyse du génome des bactéries, et plus particulièrement sur l’analyse des régions impliquées dans le codage des récepteurs membranaires de la bactérie, porte d’entrée des phages. « Nous ne sommes pas ici devant une " boîte noire ", et c’est ce qui fait la force de notre modèle à base d’IA. Nous savons exactement comment il fonctionne, ce qui nous aide à améliorer ses performances », souligne Hugo Vaysset co-premier auteur de l’article et doctorant au laboratoire Diversité moléculaire des microbes à l’Institut Pasteur. Après plus de deux ans de conception et d’entraînement, l’IA a ainsi été capable de prédire correctement l’efficacité des bactériophages face aux bactéries E. coli de la base de données dans 85 % des cas, simplement en analysant l’ADN des bactéries. « C’est un résultat qui surpasse nos attentes », avoue Aude Bernheim. Pour aller plus loin, les chercheurs ont testé leur modèle sur une nouvelle collection de souches bactériennes d’E. coli responsable de pneumonies et ont sélectionné, pour chacune d’entre elles, un « cocktail » sur mesure de trois bactériophages. Dans 90 % des cas, les bactériophages choisis sur mesure par l’IA ont réussi leur mission et détruit les bactéries en présence. Cette méthode, facilement utilisable dans les laboratoires de biologie hospitalière, ouvre la voie dans les années à venir à une sélection personnalisée et rapide de traitements par bactériophages en cas de diagnostic d’infection bactérienne à Escherichia coli très résistants aux antibiotiques. « Nous devons encore tester le comportement des phages dans différents environnements, mais la preuve de concept est faite. Nous espérons pouvoir l’étendre à d’autres bactéries pathogènes, car notre IA a été conçue pour s’adapter facilement à d’autres cas de figure, et offrir dans le futur des traitements de phagothérapie personnalisés », conclut Aude Bernheim.
(1) En France, les phages peuvent être utilisés dans le cadre d’une Autorisation temporaire d’utilisation (ATU) nominative
(2) https://www.who.int/europe/fr/news/item/25-06-2024-building-evidence-for-the-use-of-bacteriophages-against-antimicrobial-resistance
(3) Groupe de bactéries ayant des caractéristiques communes au sein d’une espèce donnée
Source :
Prediction of strain level phage–host interactions across the Escherichia genus using only genomic information, Nature Microbiology, 31 octobre 2024
Baptiste Gaborieau1,2,3,8, Hugo Vaysset4,5,6,8, Florian Tesson1,5,8, Inès Charachon1, Nicolas Dib1, Juliette Bernier1, Tanguy Dequidt3, Héloïse Georjon5, Olivier Clermont1, Pascal Hersen6, Laurent Debarbieux3, Jean-Damien Ricard1,2, Erick Denamur1,7 & Aude Bernheim5
1. Université Paris Cité, INSERM, UMR1137, IAME, Paris, France.
2. AP-HP, Hôpital Louis Mourier, DMU ESPRIT, Service de Médecine Intensive Réanimation, Colombes, France.
3. Institut Pasteur, Université Paris Cité, CNRS UMR6047, Microbiologie Intégrative et Moléculaire, Bacteriophage Bacterium Host, Paris, France.
4. AgroParisTech, Université Paris-Saclay, Paris, France.
5. Institut Pasteur, Université Paris Cité, INSERM U1284, SEED, Molecular Diversity of Microbes lab, Paris, France.
6. Institut Curie, Université PSL, Sorbonne Université, CNRS UMR168, Laboratoire Physico Chimie Curie, Paris, France.
7. AP-HP, Hôpital Bichat, Laboratoire de Génétique Moléculaire, Paris, France.
8. Ces auteurs ont contribué de manière égale : Baptiste Gaborieau, Hugo Vaysset, Florian Tesson.