La suite logicielle ARIA développée à l’Institut Pasteur automatise l’analyse des données RMN aux fins du calcul des structures tridimensionnelles des macromolécules. Pour améliorer l’expérience des utilisateurs d’ARIA tout en étendant et en simplifiant son accès, l’unité Bioinformatique structurale a créé le serveur ARIAweb, qui propose un service en ligne de calculs intensifs et offre un visualiseur moléculaire dédié ainsi que des représentations graphiques de différents scores de qualité.
Le serveur ARIAweb (https://ariaweb.pasteur.fr) fournit un accès en ligne aux fonctionnalités principales du logiciel ARIA (conversion de données RMN et calcul de structures). De plus, les structures moléculaires calculées peuvent être aisément visualisées grâce à un outil dédié et les scores de qualité sont présentés par des affichages graphiques.
Afin d’améliorer l’expérience des utilisateurs d’ARIA et fournir un accès plus large aux méthodes de calcul automatisé en RMN, l’Unité Bioinformatique structurale a développé le serveur ARIAweb (en collaboration avec le Hub Bioinformatique et Biostatistique du Département de Biologie Computationnelle), dans le but de non seulement guider les utilisateurs (du novice à l’expert) au travers des différentes étapes nécessaires au calcul de structure RMN de macromolécules, mais aussi d’offrir un service en ligne pour des calculs intensifs. Depuis 2019, ARIA est reconnu comme un service ELIXIR-EU (https://elixir-europe.org/).
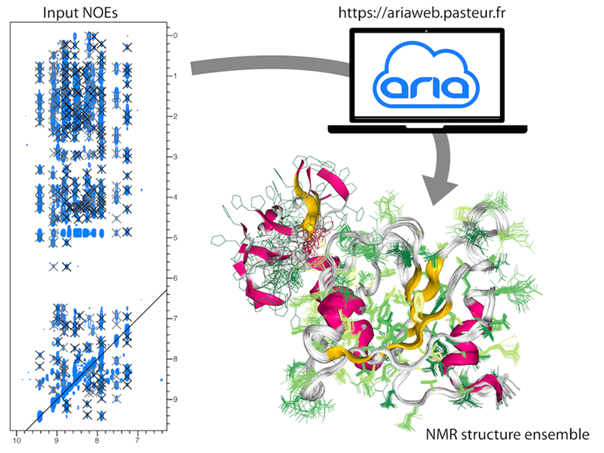
Le calcul de structure de macromécules à partir des données expérimentales RMN, simplifié grâce au serveur ARIAweb. Depuis n’importe quel navigateur Web, les utilisateurs peuvent soumettre leurs données RMN, obtenir aisément les structures calculées en ligne, et ensuite les visualiser et les analyser.
Source: Nucleic Acids Res. 2020 Jul 2. doi: 10.1093/nar/gkaa362.