La structure de l’ADN et des chromosomes est très étudiée, mais celle-ci présente encore son lot de mystère. Des chercheurs de l’Institut Pasteur et du CNRS ont développé une nouvelle méthode leur permettant de quantifier les boucles de chromatine, et d’observer les chromosomes en haute résolution.
Dans nos cellules, l’ADN est enroulé de nombreuses fois, ce qui lui permet de tenir dans le noyau cellulaire, environ 200 000 fois plus petit que la longueur de l’ADN dépliée. L’ADN est d’abord enroulé autour de protéines appelées histones, pour former une structure appelée nucléosome. Ces nucléosomes forment à leur tour une structure appelée fibre de chromatine. On sait depuis quelques années que cette fibre de chromatine forme des boucles, maintenues par un complexe de molécules appelée cohésine. Ces boucles de chromatine contribuent à structurer les chromosomes et jouent un rôle important pour les fonctions fondamentales du génome comme l’expression des gènes.
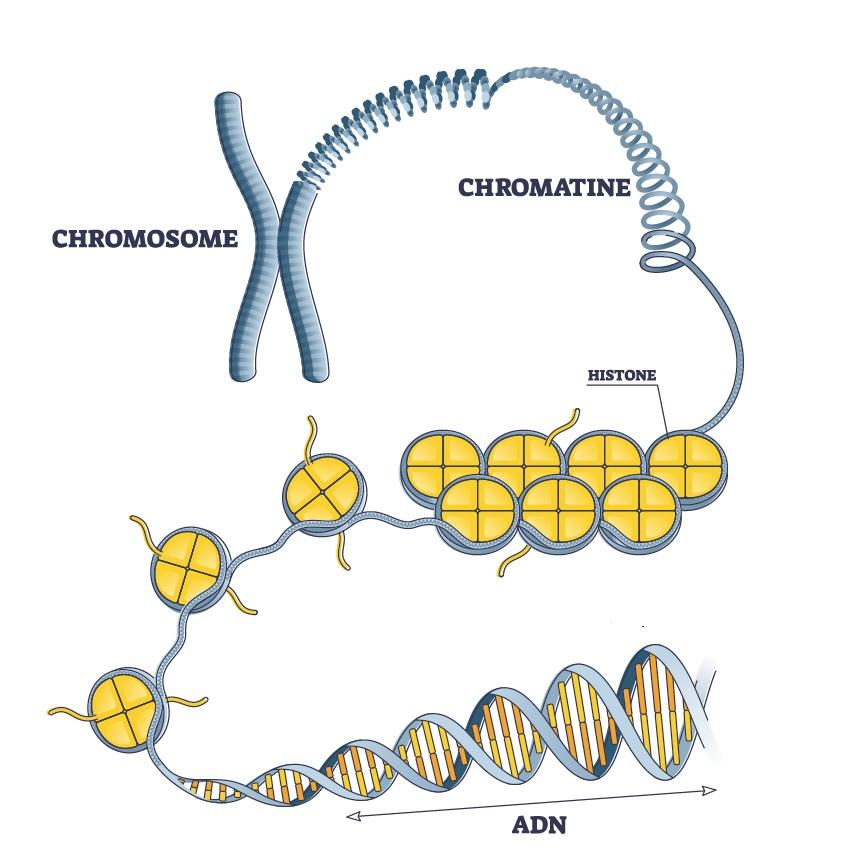
Schéma très simplifié de la compaction de l’ADN en chromosome. Le chromosome est ici symbolisé sous forme de X (métaphase). Les chercheurs de l’Institut Pasteur, dans leur article du 11 mai 2021, ont étudié l’ADN dans une autre phase du cycle cellulaire (l’interphase) où les chromosomes ont une forme différente (que l’on peut qualifier de « spaghetti ».
Une nouvelle approche pour observer la chromatine
De nos jours, l’organisation de la chromatine dans le noyau est principalement étudiée grâce à la technique Hi-C, qui utilise le séquençage pour quantifier les contacts entre différentes régions de l’ADN. « Le Hi-C fournit généralement des moyennes sur des grandes populations de cellules, et ne permet pas de bien caractériser la présence ou le nombre de ces boucles à l’échelle des cellules individuelles. Nous avons développé une approche d'imagerie combinée à des simulations de polymères pour décrire la structure de la chromatine dans des cellules humaines individuelles », explique Christophe Zimmer, responsable du laboratoire Imagerie et modélisation à l’Institut Pasteur
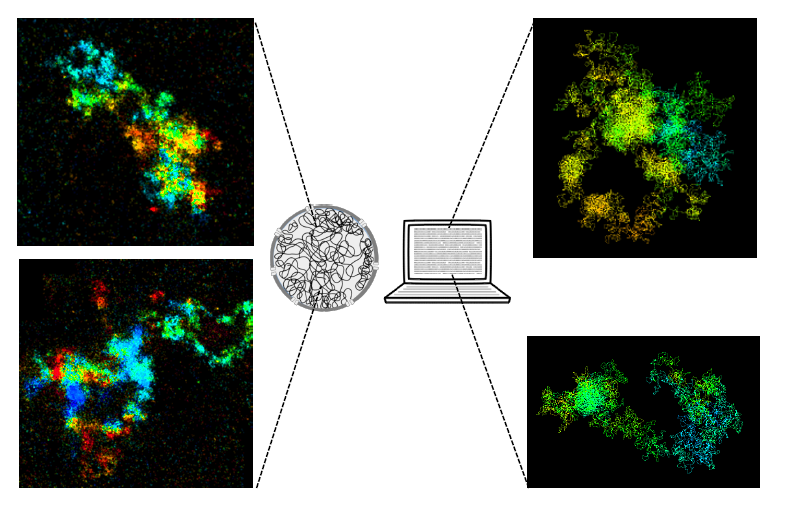
Imagerie et modélisation de chromosomes humains. A gauche, deux images de chromosomes observées avec un procédé de microscopie 3D à haute résolution. A droite, deux images de chromosomes issues d’une simulation par ordinateur.
La majorité de l’ADN humain prisonnier de boucles maintenues par la cohésine
Cette nouvelle approche permet de visualiser des chromosomes ou des régions chromosomiques en haute résolution et de façon isolée, dans des noyaux intacts et sans dénaturation de l’ADN. Les chercheurs ont utilisé cette méthode pour analyser l’influence de la cohésine sur les structures de chromatine. Leurs analyses suggèrent la présence simultanée de dizaines ou centaines de milliers de boucles dans le génome de cellules individuelles, indiquant que la grande majorité de l'ADN humain est prisonnier de boucles maintenues par la cohésine.
Cette étude fournit donc de nouvelles informations sur des structures fondamentales de l'organisation des génomes, ainsi qu'une méthode pour visualiser et modéliser la structure fine de la chromatine.
Source
Super-resolution visualization and modeling of human chromosomal regions reveals cohesin-dependent loop structures, Genome Biology, 11 mai 2021
Xian Hao1,2†, Jyotsana J. Parmar1,3†, Benoît Lelandais1†, Andrey Aristov1, Wei Ouyang1,4, Christian Weber1 and Christophe Zimmer1*
†Xian Hao, Jyotsana J. Parmar and Benoît Lelandais contributed equally to this work.
1Institut Pasteur, Imaging and Modeling Unit, UMR 3691, CNRS, Paris, France
2School of Public Health, Nanchang University, Nanchang 330006, China.
3Simons Center for the Study of Living Machines, National Center for Biological Sciences (TIFR), Bangalore, Karnataka 560065, India.
4Université Paris Diderot, PSL University, F-75005 Paris, France.