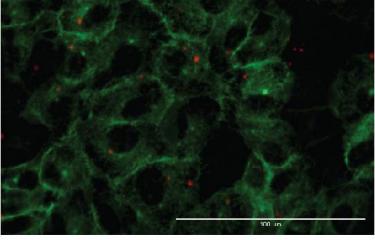
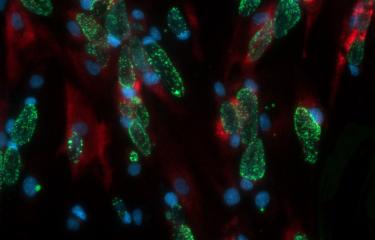
Des chercheurs de l’Institut Pasteur ont mis au point une méthode pour isoler des midbodies, ces ponts qui relient les cellules en cours de cytocinèse. En utilisant des méthodes récentes de spectrométrie de masse, ils ont dressé un inventaire exhaustif et révélé la présence de 1732 protéines ! En caractérisant trois de ces protéines en détail, ils révèlent de nouveaux mécanismes moléculaires communs à la division cellulaire et au bourgeonnement des virus.
L’unité Trafic membranaire et division cellulaire (TMDC), au sein du département Biologie Cellulaire et Infection de l’Institut Pasteur, s’intéresse à une question fondamentale de la biologie : comment une cellule donne naissance à deux cellules filles identiques ? En particulier, comment une cellule mère se coupe-t-elle physiquement en deux pour former deux cellules filles indépendantes (cytocinèse) ?
Dès 1891, le grand zoologiste allemand Walther Flemming avait décrit que les cellules en cours de cytocinèse étaient reliées par un pont intercellulaire, au centre duquel se trouve une structure morphologiquement unique appelée midbody ou Flemming body. Depuis une dizaine d’année, les chercheurs ont compris que le midbody constitue une plateforme clé permettant de recruter l’ensemble des protéines responsables de la coupure finale, ou abscission. Identifier les protéines présentes au midbody est donc une étape indispensable pour révéler de nouveaux mécanismes moléculaires responsables de l’abscission, encore largement méconnus.
Une méthode pour isoler des midbodies
L’unité TMDC vient de mettre au point une méthode pour isoler des midbodies parfaitement pur et intact, à partir de cellules humaines en culture, et d’en dresser l’inventaire exhaustif en utilisant les méthodes les plus récentes de spectrométrie de masse. Cet inventaire inédit, appelé Flemmingsome en l’honneur de W. Flemming, révèle la présence au midbody d’un nombre étonnamment important de protéines (1732). Parmi elles, 489 sont enrichies dans cette structure, dont plus de 300 pour la première fois et constituent autant de nouveaux candidats pour l’abscission. Trois protéines (Syndecan4-Syntenin-ALIX) ont été caractérisées en détail, révélant la première protéine membranaire directement impliquée dans la cytocinèse.
Ces travaux ont été conduits en lien avec d’autres équipes de recherche de l’Institut Pasteur : la plateforme de cytométrie en flux (mise au point de protocole de purification), la plateforme protéomique (mise au point de nouvelles méthodes d’isolement des peptides), le hub de bioinformatique et biostatistiques (analyse statistique et la création du site web dédié) et la plateforme d’imagerie (microscopie électronique, microscopie super-résolution).
Des avancées en biologie cellulaire fondamentale et de nouvelles perspectives pour la virologie
De façon inattendue, ce travail a également permis de révéler de nouveaux mécanismes moléculaires communs entre les étapes ultimes de la division cellulaire et le relargage de certains virus enveloppés (comme HIV, Ebola ou HBV) à partir des cellules infectées. Ces avancées ouvrent ainsi de nouvelles perspectives à la fois en biologie cellulaire fondamentale et en virologie, en proposant des cibles inédites dont l’inhibition pourrait potentiellement perturber la propagation virale.




Source
The Flemmingsome reveals an ESCRT-to-membrane coupling via ALIX/syntenin/syndecan-4 required for completion of cytokinesis, Nature Communications, 22 avril 2020.
Cyril Addi1,2, Adrien Presle1,2, Stéphane Frémont1, Frédérique Cuvelier1, Murielle Rocancourt1, Florine Milin1, Sandrine Schmutz3, Julia Chamot-Rooke4, Thibaut Douché5, Magalie Duchateau5, Quentin Giai Gianetto5,6, Audrey Salles7, Hervé Ménager6, Mariette Matondo5, Pascale Zimmermann8,9, Neetu Gupta-Rossi 1,10 & Arnaud Echard 1,10
1. Membrane Traffic and Cell Division Lab, Institut Pasteur, UMR3691, CNRS, F-75015 Paris, France.
2. Sorbonne Université, Collège doctoral, F-75005 Paris, France.
3. Institut Pasteur, UTechS CB, F-75015 Paris, France.
4. Institut Pasteur, Mass Spectrometry for Biology Unit, C2RT, USR 2000, CNRS, F-75015 Paris, France.
5. Institut Pasteur, Proteomics Platform, Mass Spectrometry for Biology, C2RT, USR 2000, CNRS, F-75015 Paris, France.
6. Hub de Bioinformatique et Biostatistique – Département Biologie Computationnelle, Institut Pasteur, USR 3756 CNRS, F-75015 Paris, France.
7. UTechS Photonic BioImaging PBI (Imagopole), Centre de Recherche et de Ressources Technologiques C2RT, Institut Pasteur, Paris 75015, France.
8. Centre de Recherche en Cancérologie de Marseille (CRCM), Equipe labellisée Ligue 2018, Aix-Marseille Université, Inserm, CNRS, Institut Paoli Calmettes, 13009 Marseille, France.
9. KU Leuven, Department of Human Genetics, University of Leuven, B-3000 Leuven, Belgium.
10. These authors jointly supervised: Neetu Gupta-Rossi, Arnaud Echard.