Deux études récentes de chercheurs de l’Institut Pasteur et de leurs collaborateurs apportent un éclairage nouveau sur l’écologie et l’évolution de la bactérie pathogène Listeria monocytogenes. Ces publications mettent en évidence la forte prévalence du portage fécal du pathogène, qui dépend du microbiote, et de la diffusion mondiale de son principal clone clinique.
Listeria monocytogenes est un pathogène humain d’origine alimentaire majeur, associé à un lourd impact médico-économique. Ce microorganisme modèle a permis des avancées majeures dans l’étude des interactions hôte-pathogène et de la physiopathologie des maladies infectieuses. Cependant, malgré son importance médicale et scientifique, L. monocytogenes n’avait pas fait l’objet d’études approfondies sur la répartition géographique de sa diversité génomique (phylogéographie) et les facteurs qui ont favorisé son émergence et sa propagation. Deux nouvelles études menées par l’Institut Pasteur en collaboration avec l'Université de Paris, l'hôpital Necker-Enfants malades AP-HP et des équipes internationales permettent aujourd’hui de préciser les niches écologiques de Listeria et la diffusion mondiale de son principal clone clinique.
Identification des niches écologiques de L. monocytogenes
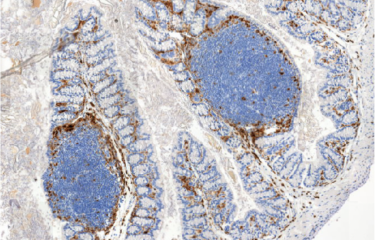
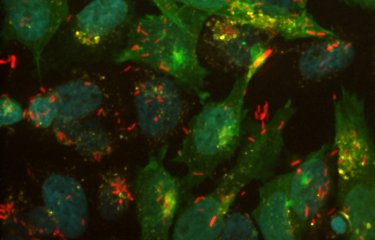
Dans une étude publiée dans Nature Communications, l’unité Biologie des infections de l’Institut Pasteur et de l’Inserm, associée à des chercheurs des universités de Lausanne et de Poitiers ont examiné la répartition des espèces de Listeria en exploitant plus de 10 000 ensembles de données métagénomiques. Ils ont montré que L. monocytogenes était l’espèce du genre Listeria la plus prévalente, qu’elle était principalement associée à divers hôtes – humains, bovins et autres animaux d’élevage – et qu’elle était également présente dans le sol. Ils ont aussi constaté que les autres espèces de Listeria non virulentes étaient quant à elles pratiquement absentes de ces mêmes hôtes.
De plus, l’analyse d’une cohorte indépendante de près de près de 1 000 échantillons fécaux de donneurs asymptomatiques a révélé que la prévalence du portage fécal asymptomatique de L. monocytogenes est d’environ 10%, ce qui suggère l’implication de la pathogénicité de L. monocytogenes dans son pouvoir d’association à l’hôte, même en l’absence de symptômes. L’analyse plus approfondie des jeux de données métagénomiques indique que le portage fécal de L. monocytogenes est associé à la nature du microbiote. Celle-ci précède la colonisation par L. monocytogenes chez un modèle de souris in vivo, démontrant que le portage fécal de L. monocytogenes dépend de la composition du microbiote intestinal. Ces découvertes démontrent que le portage fécal de L. monocytogenes est une conséquence cruciale mais jusqu’ici négligée de sa virulence.
Reconstruction de l’histoire de la transmission du pathogène
Un autre article, publié dans Science advances, souligne l’importance de l’association de L. monocytogenes à ses hôtes dans sa propagation mondiale. Il s’agit de la première étude phylogéographique de L. monocytogenes. Elle s’interesse au groupe clonal hypervirulent le plus répandu cliniquement dans les pays occidentaux : CC1. L’unité Biologie des infections de l’Institut Pasteur et l’Inserm, en collaboration avec les unités Biodiversité et épidémiologie des bactéries pathogènes et Modélisation mathématique des maladies infectieuses de l’Institut Pasteur et un consortium international, ont utilisé près de 2 000 génomes provenant de 40 pays pour retracer l’histoire évolutive de ce clone.
Ce travail révèle que le principal clone clinique CC1 est originaire d’Amérique du Nord et qu’il s’est répandu dans le monde entier après la révolution industrielle, à raison de deux vagues successives d’expansion. Cette propagation a coïncidé d’une part avec l’essor du commerce transatlantique de viande et de bovins au cours de la seconde moitié du 19e siècle, et d’autre part avec la croissance rapide de l’élevage bovin et des filières alimentaires au 20e siècle. Fait intéressant, les chaînes de transmission qui étaient mondiales au cours du siècle dernier sont désormais principalement locales, dans l’environement des fermes ou des sites de production alimentaires, et tendent à persister longtemps, avec une diffusion limitée entre les pays.
Ces deux études fournissent une vue d’ensemble des réservoirs de L. monocytogenes et de la dynamique de sa transmission. Elles soulignent le rôle central joué par le bétail dans la propagation et l’évolution de ce pathogène alimentaire majeur. Leurs résultats mettent également en évidence la nécessité de renforcer la surveillance des animaux afin de limiter la transmission de L. monocytogenes à l’Homme par la chaîne alimentaire.
Pour plus d’informations :
Centre collaborateur de l’OMS : sur Pasteur.fr / sur site web de l'OMS (en anglais)
Cette étude entre dans le cadre de l’axe scientifique prioritaire Maladies infectieuses émergentes du plan stratégique 2019-2023 de l’Institut Pasteur.
Sources:
Listeria monocytogenes faecal carriage is common and depends on the gut microbiota, Nature Communications, 24 novembre 2021
Lukas Hafner1, Maxime Pichon2,3,, Christophe Burucoa2,3,, Sophie H. A. Nusser1, Alexandra Moura1,4, Marc Garcia-Garcera5 and Marc Lecuit1,4,6
1 - Institut Pasteur, Université de Paris, Inserm U1117, Biology of Infection Unit, 75015 Paris, France.
2 - University Hospital of Poitiers, Infectious Agents Department, Bacteriology and Infection Control Laboratory, 86021 Poitiers, France.
3 - Université de Poitiers, Faculté de Médecine et de Pharmacie, EA 4331, 86022 Poitiers, France.
4 - Institut Pasteur, National Reference Center and WHO Collaborating Center Listeria, 75015 Paris, France.
5 - University of Lausanne, Department of Fundamental Microbiology, 1015 Lausanne, Switzerland.
6 - Necker-Enfants Malades University Hospital, Division of Infectious Diseases and Tropical Medicine, APHP, Institut Imagine, 75006 Paris, France.
Emergence and global spread of Listeria monocytogenes main clinical clonal complex, Science Advances, 1er décembre 2021
Alexandra Moura1,2, Noémie Lefrancq3, Thierry Wirth4,5, Alexandre Leclercq1,2, Vítor Borges6, Brent Gilpin7, Timothy J. Dallman8, Joachim Frey9, Eelco Franz10, Eva M. Nielsen11, Juno Thomas12, Arthur Pightling13, Benjamin P. Howden14,15, Cheryl L. Tarr16, Peter Gerner-Smidt16, Simon Cauchemez3, Henrik Salje3, Sylvain Brisse17, Marc Lecuit1,2,18
1 - Institut Pasteur, Université de Paris, Inserm U1117, Biology of Infection Unit, Paris, France.
2 - Institut Pasteur, National Reference Center and WHO Collaborating Center Listeria, 75015 Paris, France.
3 - Institut Pasteur, Université de Paris, Mathematical Modelling of Infectious Diseases Unit, CNRS UMR 2000, Paris, France.
4 - Institut Systématique Evolution Biodiversité (ISYEB),Museum National d’Histoire Naturelle,
CNRS, Sorbonne Université, Université des Antilles, EPHE, Paris, France.
5 - PSL University, EPHE, Paris, France.
6 - Department of Infectious Diseases, National Institute of Health Dr. Ricardo Jorge, Lisbon, Portugal.
7 - Christchurch Science Centre, Institute of Environmental Science and Research Limited, Christchurch, New Zealand.
8 - Public Health England, London, UK.
9 - Vetsuisse Faculty, University of Bern, Bern, Switzerland.
10 - Centre for Infectious Disease Control, National Institute for Public Health and the Environment (RIVM), Bilthoven, Netherlands.
11 - Statens Serum Institut, Copenhagen, Denmark.
12 - Division of the National Health Laboratory Service, National Institute for Communicable Diseases, Johannesburg, South Africa.
13 - Biostatistics and Bioinformatics, Center for Food Safety and Applied Nutrition, U.S. Food and Drug Administration, College Park, MD, USA.
14 - Microbiological Diagnostic Unit Public Health Laboratory, Department of Microbiology and Immunology, The Doherty Institute for Infection ,and Immunity, University of Melbourne, Melbourne, Victoria, Australia.
15 - Infectious Diseases Department, Austin Health, Heidelberg, Victoria, Australia.
16 - Centers for Disease Control and Prevention, Atlanta, GA, USA.
17 - Institut Pasteur,Université de Paris, Biodiversity and Epidemiology of Bacterial Pathogens Unit, Paris, France.
18 - Necker-Enfants Malades University Hospital, Division of Infectious Diseases and Tropical Medicine, APHP, Institut Imagine, Paris, France.