La calmoduline est une protéine naturellement produite par l’organisme et dont l’activité est modulée par le calcium à l’intérieur de nos cellules. Des chercheurs de l’Institut Pasteur ont réussi à préciser les mécanismes d’interaction entre la calmoduline et le calmidazolium. Ce dernier peut diminuer efficacement les effets biologiques de la calmoduline, mais est également toxique pour les cellules. Cette étude ouvre la voie au développement de nouveaux inhibiteurs plus sélectifs de la calmoduline avec moins d’effets indésirables.
La calmoduline est une protéine présente dans tous les organismes vivants, du moins chez ceux dont les cellules comportent un noyau. « Calmoduline » signifie littéralement protéine « qui est modulée par le calcium », un élément clé du métabolisme cellulaire. De ce fait, la calmoduline est impliquée dans des fonctions biologiques variées : architecture et mouvement des cellules, division cellulaire, communication entre cellules… Elle est aussi impliquée dans des pathologies comme les troubles du rythme cardiaque, l’évolution de la maladie d’Alzheimer…. La calmoduline est également détournée de ses fonctions par des toxines bactériennes dans les processus d’intoxication cellulaire (cf. Mécanisme d’activation d’une toxine majeure impliquée dans la coqueluche – L’actu de l’Institut Pasteur). Agir sur la calmoduline est donc un sujet important en recherche fondamentale et en santé.
Calcium, calmoduline, calmidazolium : des interactions mal connues
On sait déjà qu’en se liant à la calmoduline, le calcium en modifie la conformation moléculaire. La calmoduline devient alors un « interrupteur » protéique qui régule tout un ensemble d’autres protéines. Malgré son importance dans le fonctionnement cellulaire, sa structure et sa dynamique constituent encore un sujet d’intenses débats et de recherches.
Un des antagonistes de la calmoduline le plus étudié est le calmidazolium, découvert en 1981. Il peut se fixer très facilement à la calmoduline pour en inhiber les effets physiologiques. « Le calmidazolium, comme de nombreux autres antagonistes de la calmoduline, est malheureusement connu pour sa cytotoxicité et pour interagir avec d’autres cibles cellulaires », précise Alexandre Chenal, responsable du groupe BiophysiCyaA. Ce chercheur a déjà travaillé sur les liens entre calmoduline et la toxine CyaA, un facteur de virulence majeur de la coqueluche, au sein de l’unité Biochimie des Interactions Macromoléculaires dirigée par Daniel Ladant à l’Institut Pasteur. Personne jusqu’alors n’a réussi à résoudre la structure du complexe calmoduline-calmidazolium. Les mécanismes responsables de cette affinité sont eux aussi mal définis, et les données varient d’une publication à l’autre. L’équipe d’Alexandre Chenal a employé différentes approches technologiques pour répondre à ces problématiques.
Des technologies de pointe pour mesurer les effets du calmidazolium
Les chercheurs ont employé une technique nommée SR-CD, au synchrotron SOLEIL. « Nous avons pu ainsi démontrer que le calmidazolium ne modifie pas les repliements locaux de la calmoduline », explique Corentin Léger, post-doctorant dans le groupe de recherche BiophysiCyaA et premier auteur de la publication.
Grâce à une méthode de quantification (dite ITC, avec la plateforme de Biophysique moléculaire de l’Institut Pasteur), les chercheurs ont aussi précisé l’affinité de l’interaction entre la calmoduline et le calmidazolium, et ont montré que la liaison d’un seul calmidazolium suffit pour inhiber la calmoduline. Il s’agissait ensuite de déterminer si l’interaction entre la calmoduline et son inhibiteur modifie la conformation spatiale de la calmoduline.
Les chercheurs ont alors employé d’autres technologies d’analyse diversifiées :
- Le SEC-SAXS (Chromatographie d’Exclusion Stérique – Diffusion de rayons X aux petits angles), pour étudier la conformation spatiale du complexe calmoduline-calmidazolium ; une technologie du synchrotron SOLEIL ;
- Les méthodes de spectrométrie de masse HDX-MS (échange hydrogène-deutérium) et de résonance magnétique nucléaire (RMN), pour mieux caractériser le complexe calcium-calmoduline ; deux technologies d’une plateforme technologique de l’Institut Pasteur.
Les chercheurs ont ainsi mis en évidence qu’en présence de calmidazolium, la calmoduline initialement étendue et flexible prenait une conformation compacte et rigide. Le calmidazolium agit comme une « glue » au cœur de la molécule, ce qui maintient la calmoduline dans une conformation fermée (cf. figure ci-dessous). Elle n’est alors plus disponible pour interagir avec ses partenaires physiologiques.
Grâce une technique de cristallographie basée sur les rayons X (avec la plateforme de cristallographie et le synchrotron SOLEIL), les chercheurs ont réussi à déterminer la structure atomique du complexe formé par la calmoduline et le calmidazolium. Par ailleurs, les expériences de RMN et de HDX-MS ont montré que la liaison du calmidazolium réduit la dynamique de la calmoduline, et la fige dans une conformation compacte et fermée (cf figure).
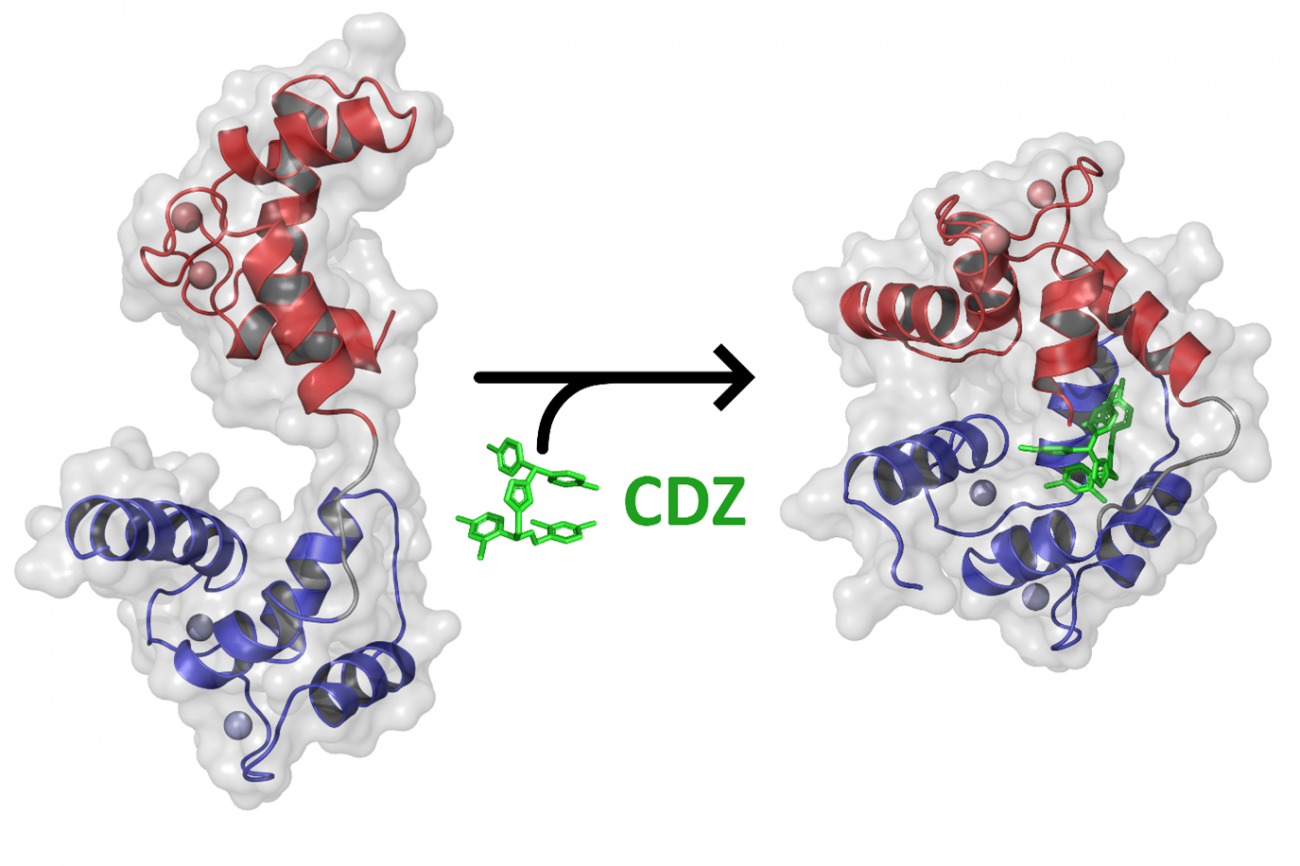
L’ajout d’un excès de calmidazolium ne change pas la structure globale du complexe calmoduline-calmidazolium. Ainsi, un seul calmidazolium est suffisant pour changer la conformation de la calmoduline et pour l’inhiber.
Comme évoqué plus haut, le calmidazolium ne se fixe pas uniquement sur la calmoduline : il interagit également avec d’autres composants cellulaires, provoquant des effets indésirables. Ces travaux devraient permettre aux chimistes et bio-informaticiens de développer de nouveaux inhibiteurs plus spécifiques de la calmoduline et moins toxiques pour les cellules. « Ces nouvelles molécules seront utiles en recherche fondamentale pour l’étude de la calmoduline et, éventuellement à long terme, pourraient être des candidats pour la thérapie de pathologies impliquant la calmoduline », conclut Alexandre Chenal.
Cette étude illustre également les synergies entre les équipes de recherche de l’Institut Pasteur et ses plateformes technologiques, notamment la plateforme de biophysique moléculaire, celles de cristallographie et de RMN biologique, ainsi que les lignes DISCO, SWING et PROXIMA du synchrotron SOLEIL (Saint-Aubin, France).
Source
Dynamics and structural changes of calmodulin upon interaction with its potent antagonist Calmidazolium, BMC Biology, 09 août 2022
Corentin Léger1, Irène Pitard2, Mirko Sadi1,3, Nicolas Carvalho1,3, Sébastien Brier2, Ariel Mechaly4, Dorothée Raoux-Barbot1, Maryline Davi1, Sylviane Hoos5, Patrick Weber4, Patrice Vachette6, Dominique Durand6, Ahmed Haouz4, J. Iñaki Guijarro2, Daniel Ladant1, Alexandre Chenal1
1: Biochemistry of Macromolecular Interactions Unit, Department of Structural Biology and Chemistry, Institut Pasteur, Université de Paris Cité, CNRS UMR3528, 75015 Paris, France
2: Biological NMR and HDX-MS Technological Platform, Institut Pasteur, Université de Paris Cité, CNRS UMR3528, 75015 Paris, France
3: Université de Paris Cité, Sorbonne Université, Paris, France
4: Plate-forme de Cristallographie-C2RT, Institut Pasteur, Université de Paris Cité, CNRS UMR3528, Paris, France
5: Plateforme de Biophysique Moléculaire, Institut Pasteur, Université de Paris Cité, CNRS UMR3528, Paris, France
6: Université Paris-Saclay, CEA, CNRS, Institute for Integrative Biology of the Cell (I2BC), 91198, Gif-sur-Yvette, France