Des chercheurs de l’Institut Pasteur et du CNRS, en collaboration avec des équipes américaines et australiennes, ont réalisé une caractérisation génomique et évolutive complète de la souche de bactérie Elizabethkingia anophelis à l’origine d’une épidémie qui a touché une soixantaine de personnes dans le Wisconsin en 2015-2016. L’étude révèle que la souche d’E. anophelis à l’origine de cette épidémie est fortement mutante. Une propriété inhabituelle pour une bactérie, qui pourrait conférer un avantage sélectif à cette souche par ailleurs exceptionnellement résistante à de nombreux antibiotiques. Ces données, publiées le 24 mai 2017 dans la revue Nature Communications, ont été mises à disposition de la communauté scientifique internationale.
En novembre 2015 s’est déclarée dans le Wisconsin (Etats-Unis) une épidémie causée par la bactérie Elizabethkingia anophelis, peu connue jusqu’alors. Au total, 66 cas ont été confirmés dans le pays : une ampleur exceptionnelle pour une épidémie due à cette bactérie. Malgré la mise en place un protocole de traitement antibiotique, 21 patients sont décédés, la majorité âgés de plus de 65 ans et présentant au moins une pathologie sérieuse sous-jacente.
Afin de comprendre cette épidémie hors norme, les chercheurs de l’unité de Génomique évolutive des microbes (Institut Pasteur/CNRS), de l’Institut français de bioinformatique (CNRS/Inserm/Inra/Inria/CEA) et du Centre de bioinformatique, biostatistique et biologie intégrative (Institut Pasteur/CNRS), menés à l’Institut Pasteur par Sylvain Brisse, en collaboration avec des équipes du Center for Disease Control and prevention, du Wisconsin Department of Health Services, du Wisconsin State Laboratory of Hygiene (Etats-Unis) et de l’Université de Melbourne (Australie) ont mené un important travail d’investigation génomique sur les isolats issus de patients de cet épisode épidémique.
Les chercheurs français avaient déjà décrit plusieurs lignées de cette bactérie dans le cadre d’une précédente épidémie en République centrafricaine et comparé les données à des génomes de banques biologiques publiques. Ces travaux leur avaient alors permis de définir un système de typage standardisé, qu’ils ont pu appliquer à la caractérisation de la souche en cause dans l’épidémie du Wisconsin.
 |
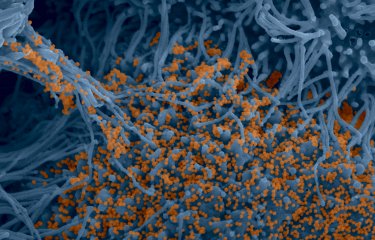
Elizabethkingia anophelis est une bactérie décrite pour la première fois en 2011 à partir du moustique anophèle mais il n’y a aucune indication d’un rôle du moustique dans sa transmission à l’Homme. C’est plutôt une contamination environnementale, par l’eau par exemple, qui est suspectée. Les bactéries Elizabethkingia causent très rarement des infections chez l’Homme mais peuvent éventuellement coloniser l’appareil respiratoire. En cas d’infection les symptômes peuvent inclure de la fièvre, des difficultés à respirer et des frissons. E. anophelis a été précédemment impliquée dans deux cas de méningites néonatales en Centrafrique et dans une micro-épidémie de six cas d’infections dans une unité de soins intensifs à Singapour. © CDC special bacteriology reference lab. |
Le séquençage des isolats bactériens, réalisé par le CDC d’Atlanta, a en premier lieu permis de déterminer que l’épidémie était « clonale », c’est-à-dire qu’elle n’était due qu’à une seule souche d’E. anophelis. L’ensemble des cas d’infections de l’épidémie représente ainsi une nouvelle lignée dans l’évolution d’ E. anophelis. A noter que l’épidémie s’est déclenchée de manière communautaire, c’est-à-dire à l’extérieur des structures de soins, ce qui est inhabituel, les infections à Elizabethkingia ayant toujours été associées aux soins jusqu’alors.
Mais l’étude a surtout révélé le caractère hautement mutant de la souche incriminée. « Nous avons montré que la lignée avait accumulé près de 300 mutations en un an. C’est 10 fois plus qu’une bactérie « classique ». Cette mutabilité est extrêmement rare pour les épidémies à bactéries », commente Sylvain Brisse. Les scientifiques ont plus précisément déterminé que cette propriété s’expliquait par l’insertion d’un élément génétique mobile dans le génome de la bactérie au niveau d’un gène de réparation de l’ADN.
Bien que ces nombreuses mutations accumulées n’aient pas directement rendu la souche plus résistante aux antibiotiques – E. anophelis est déjà connue pour être très résistante –, les chercheurs font l’hypothèse que cette capacité mutante a conféré un avantage sélectif à la bactérie pour sa survie dans le réservoir environnemental, ou dans la source infectieuse. Elle a également pu augmenter son pouvoir pathogène chez l’homme. Les scientifiques ont par ailleurs identifié d’autres caractéristiques particulières comme des gènes de détoxification ou d’utilisation de substances organiques végétales. Ces derniers pourraient avoir permis à la bactérie de coloniser plus efficacement le réservoir environnemental et contribuer alors à expliquer l’ampleur de l’épidémie.
Ces observations ont amené les chercheurs à retracer la diversification des bactéries pendant l’épidémie. Ils ont ainsi daté l’apparition de leur ancêtre commun à un an avant le premier cas d’infection confirmé. « Cette information a donné une profondeur temporelle additionnelle à l’évènement, ce qui a permis de nouvelles hypothèses épidémiologiques et aurait pu conduire à identifier a posteriori de nouveaux cas », poursuit le chercheur.
Au-delà de la découverte d’une bactérie mutante responsable d’une épidémie, cette étude a été l’occasion pour les chercheurs de mettre en avant l’intérêt de l’échange de données en temps réel pendant les épidémies, en accord avec le mouvement de « l’open data », pour mieux faire face aux urgences de santé publique.
Ce travail a reçu le soutien financier de l’Institut Pasteur, du CNRS, de la Fondation pour la Recherche Médicale, de l’Institut Français de Bioinformatique, du LabEx IBEID (The laboratory of excellence Biologie Intégrative des maladies infectieuses émergentes, géré par l’Agence Nationale de Recherche), et du Advanced Molecular Detection (AMD) initiative du CDC (Atlanta, USA).
Source
Evolutionary dynamics and genomic features of the Elizabethkingia anophelis 2015 to 2016 Wisconsin outbreak strain, Nature Communications, 24 mai 2017.
Amandine Perrin (a,b,c)&, Elise Larsonneur (a,b,d)&, Ainsley C. Nicholson (e)&, David J. Edwards (f,g), Kristin M. Gundlach (h), Anne M. Whitney (e), Christopher A. Gulvik (e), Melissa E. Bell (e), Olaya Rendueles (a,b), Jean Cury (a,b), Perrine Hugon (a,b), Dominique Clermont (i), Vincent Enouf(j), Vladimir Loparev (k), Phalasy Juieng (k), Timothy Monson (h), David Warshauer (h), Lina I Elbadawi (l, m), Maroya Spalding Walters (n), Matthew B Crist (n), Judith Noble-Wang (n), Gwen Borlaug (m), Eduardo P.C. Rocha (a,b), Alexis Criscuolo (c,j), Marie Touchon (a,b), Jeffrey P. Davis (m), Kathryn E. Holt (f,g), John R. McQuiston (e),*, Sylvain Brisse (a,b,o),*
a) Institut Pasteur, Microbial Evolutionary Genomics, F-75724, Paris, France
b) CNRS, UMR 3525, F-75724, Paris, France
c) Institut Pasteur, Hub Bioinformatique et Biostatistique, C3BI, USR 3756 IP CNRS, F-75724, Paris, France
d) CNRS, UMS 3601 IFB-Core, F- 91198, Gif-sur-Yvette, France
e) Special Bacteriology Reference Laboratory, Bacterial Special Pathogens Branch, Division of High Consequence Pathogens and Pathology, Centers for Disease Control and Prevention, Atlanta, Georgia 30329, USA
f) Centre for Systems Genomics, University of Melbourne, Parkville, Victoria 3010, Australia
g) Department of Biochemistry and Molecular Biology, Bio21 Molecular Science and Biotechnology Institute, University of Melbourne, Parkville, Victoria 3010, Australia
h) Wisconsin State Laboratory of Hygiene, Madison, Wisconsin 53718, USA
i) CIP - Collection de l'Institut Pasteur, Institut Pasteur, F-75724, Paris, France.
j) Institut Pasteur, Pasteur International Bioresources network (PIBnet), Plateforme de Microbiologie Mutualisée (P2M), F-75724, Paris, France
k) Division of Scientific Resources, Centers for Disease Control and Prevention, Atlanta, Georgia 30329, USA
l) Epidemic Intelligence Service, Centers for Disease Control and Prevention, Atlanta, Georgia 30329, USA
m) Division of Public Health, Wisconsin Department of Health Services, Madison, Wisconsin 53701, USA
n) Division of Healthcare Quality Promotion, Centers for Disease Control and Prevention, Atlanta, Georgia 30329, USA
o) Institut Pasteur, Molecular Prevention and Therapy of Human Diseases, F-75724, Paris, France
& Les contributions de ces auteurs sont de même niveau.
* Auteurs à qui adresser la correspondance sur l’article