Trois ans après la publication de la structure du premier chromosome synthétique de levure, le consortium international Sc2.0, constitué de plus de 200 chercheurs dans le monde, annonce avoir synthétisé cinq nouveaux chromosomes, totalisant 30 % du génome du microorganisme. Ces découvertes ont fait l’objet de sept publications conjointes dans la revue Science du 10 mars 2017. Des chercheurs de l’Institut Pasteur, du CNRS et de l’université Pierre-et-Marie-Curie étudient dans l’une d’elle l’organisation fonctionnelle de ces chromosomes synthétiques. Leurs travaux permettent de valider l’ensemble du design des séquences synthétiques et serviront de référence dans des expériences futures.

Le challenge d’un tel programme réside à la fois dans la taille des séquences assemblées – des chromosomes entiers, et non pas des gènes unitaires – et dans le design de ces chromosomes, qui intègre de nombreuses modifications visant à garantir la stabilité du génome et sa fonctionnalité. Une équipe de recherche, constituée de chercheurs de l’Institut Pasteur, du CNRS et de l’université Pierre-et-Marie-Curie, a dans ce cadre coordonné le volet essentiel d’analyse de l’architecture 3D de l’ensemble de ces chromosomes synthétiques, soit individuellement, soit pour certains d’entre eux combinés dans la même souche. Leurs travaux font l’objet de l’un de ces sept articles.
L’architecture 3D des chromosomes joue un rôle important dans leur régulation fonctionnelle. Elle influence par exemple l’expression de certains gènes, ou varie au cours du cycle pour faciliter la ségrégation des chromosomes lors de la division cellulaire. L’intégrité de la structure tridimensionnelle du génome des souches de levure contenant un ou plusieurs chromosomes synthétiques était donc essentielle pour valider le design Sc2.0.
Dans leur étude, les scientifiques français révèlent ainsi que malgré l’élimination de séquences particulières (éléments répétés ou ARN de transfert) et l’introduction de milliers de mutations ponctuelles induites pour le design des chromosomes synthétiques, l’architecture de ces derniers n’est pas modifiée, et que les fonctions biologiques de la levure ne sont pas altérées.
Les molécules d’ADN synthétiques présentent en effet les mêmes caractéristiques organisationnelles que les séquences naturelles. Des expériences complémentaires montrent également que des changements d’organisation drastiques peuvent être obtenus lorsque certaines régions spécifiques sont déplacées d’un chromosome à l’autre, sans tuer ni affecter pour autant les fonctions biologiques de la levure, soulignant la robustesse de cet organisme.
Ces résultats ont été obtenus grâce à la méthode dite « Hi-C », qui quantifie les contacts physiques entre des centaines de positions le long des chromosomes dans une population de cellules. Les scientifiques ont ainsi pu accéder à l’organisation moyenne des chromosomes, puis transformer ces données en structures tridimensionnelles, facilitant leur interprétation.
Cette étude permet ainsi de valider l’avancée du projet en confirmant que l’organisation fonctionnelle des chromosomes synthétiques n’apparait pas affectée, malgré les nombreuses mutations qu’ils portent. En outre, ces travaux mettent également en évidence l’adaptabilité génomique et la robustesse de la levure Saccharomyces cerevisiae, deux qualités qui en font un modèle de choix pour le développement d’applications biotechnologiques ou biomédicales et la désignent comme une excellente plateforme pour l'ingénierie chromosomique à grande échelle. Les structures chromosomiques caractérisées dans le cadre de ce consortium pourront enfin servir de références aux analyses futures impliquant l’utilisation de ces chromosomes.
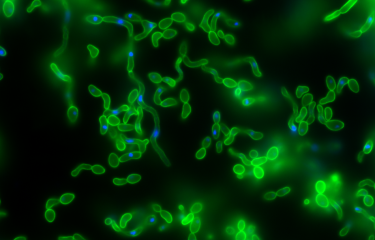
Iconographie : organisation 3D moyenne des 16 chromosomes de la levure de boulangerie. Les centromères des chromosomes sont co-localisés dans l'espace nucléaire (en haut). En doré : les cinq nouveaux chromosomes synthétiques. © Institut Pasteur
Source
3D organization of synthetic and scrambled chromosomes, Science, 10 mars 2017.
Guillaume Mercy1,2,3†, Julien Mozziconacci4†, Vittore F. Scolari1,2, Kun Yang5,Guanghou Zhao9, Agnès Thierry1,2, Yisha Luo12, Leslie A. Mitchell6, Michael Shen6, Yue Shen10,11,12, Roy Walker12, Weimin Zhang9, Yi Wu7, Ze-xiong Xie7, Zhouqing Luo9, Yizhi Cai12, Junbiao Dai9, Huanming Yang8,10, Ying-Jin Yuan7, Jef D. Boeke6, Joel S. Bader5, Héloïse Muller1,2,13*, Romain Koszul1,2*
1 Institut Pasteur, Département Génomes et génétiques, unité Régulation spatiale des génomes, Paris, 75015, France.
2 UMR3525, Centre National de la Recherche Scientifique, Paris, 75015, France.
3 Sorbonne Universités, UPMC Université Paris 6, 75005, Paris, France.
4 Laboratoire de Physique Théorique de la Matière Condensée, CNRS UMR 7600, Université Pierre et Marie Curie, Sorbonne Universités, Paris, France.
5 Department of Biomedical Engineering and High-Throughput Biology Center, Whiting School of Engineering, Johns Hopkins University, Baltimore, MD 21218, USA.
6 Institute for Systems Genetics and Department of Biochemistry & Molecular Pharmacology, NYU Langone Medical Center, New York, NY 10016.
7 Key Laboratory of Systems Bioengineering (Ministry of Education), SynBio Research Platform, Collaborative Innovation Center of Chemical Science and Engineering (Tianjin), School of Chemical Engineering and Technology, Tianjin University, Tianjin, 300072, PR China.
8 James D. Watson Institute of Genome Sciences, Hangzhou 310058, China.
9 Key laboratory for Industrial Biocatalysis (Ministry of Education), Key laboratory of Bioinformatics (Ministry of Education), Center for Synthetic and Systems Biology, School of Life Sciences, Tsinghua University, Beijing 100084, China.
10 BGI-Shenzhen, Shenzhen 518083, China.
11 BGI-Qingdao, Qingdao 266555, China.
12 School of Biological Sciences, University of Edinburgh, Edinburgh EH9 3BF, UK.
13 Adresse actuelle : Institut Curie, UMR3664 Dynamique du Noyau, Paris, France.
† Contribution de même importance de ces auteurs.