Dans un article paru dans PLoS Genetics le 5 juillet, des chercheurs de l’Institut Pasteur et de l’Université Catholique de Louvain (Belgique) décryptent pour la première fois les mécanismes génétiques et métaboliques qui sous-tendent l’action d’un bactériophage connu pour son potentiel thérapeutique. Face à l’inquiétante augmentation des résistances bactériennes aux antibiotiques et à la difficulté de concevoir de nouvelles molécules efficaces, la phagothérapie, qui repose sur l’utilisation de ces virus bactériens, connaît depuis quelques années un regain d’intérêt de la communauté scientifique. Cette découverte contribue ainsi aux efforts entrepris pour permettre le développement de ce traitement, aujourd’hui délaissé dans le monde occidental.

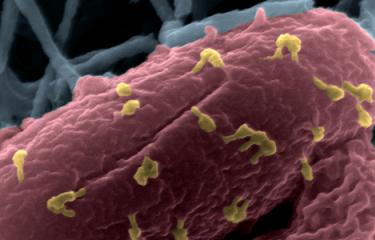
Bactériophages sur la bactérie Escherichia coli © Institut Pasteur
La phagothérapie est une approche médicale utilisée pour traiter les maladies infectieuses d’origine bactérienne, s’appuyant sur les capacités naturelles de certains virus, appelés bactériophages, à tuer les bactéries qu’ils reconnaissent spécifiquement. La phagothérapie fût proposée il y a presqu’un siècle à l’Institut Pasteur par le chercheur franco-canadien Félix d’Herelle. Bien qu’initialement utilisée dans le monde entier, à partir des années 1940 elle connut un fort essor dans les pays de l’Est, alors qu’elle fût progressivement abandonnée dans les pays occidentaux au profit des antibiotiques, jugés plus prometteurs. Aujourd’hui, la nature virale de ce traitement anti-bactérien et l’absence de connaissances précises sur le mode d’action des bactériophages constituent des obstacles au recours à la phagothérapie dans un cadre médical.
Pourtant, face à l’augmentation des résistances bactériennes aux antibiotiques et à l’inquiétante perspective des impasses thérapeutiques qui l’accompagne, des scientifiques se tournent à nouveau vers la piste des bactériophages. C’est le cas de chercheurs de l’Institut Pasteur, menés par Laurent Debarbieux[1], et de l’Université Catholique de Louvain (Belgique), menés par Rob Lavigne. Ils publient aujourd’hui une étude attendue décryptant au niveau moléculaire les mécanismes d’action originaux utilisés par un bactériophage, connu pour son potentiel thérapeutique, infectant la bactérie pathogène opportuniste Pseudomonas aeruginosa.
Les travaux des scientifiques révèlent que toute la stratégie d’infection du bactériophage, depuis le piratage de la cellule jusqu’à son propre programme de multiplication, repose sur le métabolisme de l’ARN, un mécanisme fondamental au cœur du processus de production de la matière biologique.
Les chercheurs ont en effet d’abord montré qu’au cours de son cycle infectieux, le phage était capable de déstabiliser fortement la cellule, en dégradant précocement les ARN qu’elle produit. A l’inverse, la synthèse de l’ARN du bactériophage, elle, est particulièrement active, traduisant une forte activité liée à sa propre multiplication.
En outre, les chercheurs ont également montré que le bactériophage faisait appel à des mécanismes de contrôle impliquant des fragments d’ARN appelés « petits ARN » et « ARN antisens », pour orchestrer finement les différentes étapes de production des nouvelles particules virales.
Ces travaux soulignent l’intérêt que peut présenter la caractérisation de bactériophages aux propriétés thérapeutiques, à la fois en tant que source de nouvelles découvertes fondamentales, mais également pour mieux comprendre le mode d’action des bactériophages dans leur activité antibactérienne. « Ces travaux devraient inciter à la caractérisation d’autres bactériophages à potentiel thérapeutique, commente Laurent Debarbieux. Nous espérons qu’ils contribueront, à terme, à rendre de nouveau possible en France l’utilisation de ces virus à des fins médicales ».
[1] chef du groupe Interactions Bactériophages Bactéries chez l’Animal à l’Institut Pasteur.
Source
Next-Generation“-omics”Approaches Reveal a Massive Alteration of Host RNA Metabolism during Bacteriophage Infection of Pseudomonas aeruginosa, Plos Genetics, 5 juillet 2016.
Anne Chevallereau (1,2☯),Bob G. Blasdel (3,☯),Jeroen De Smet (3), Marc Monot (4), Michael Zimmermann (5), Maria Kogadeeva (5), Uwe Sauer (5), , Peter Jorth (6), Marvin Whiteley (6), Laurent Debarbieux (1)*, Rob Lavigne (3)*
1) Institut Pasteur, Molecular Biology of the Gene in Extremophiles Unit, Department of Microbiology, Paris, France,
2) Université Paris Diderot, Sorbonne Paris Cité, Cellule Pasteur, Paris, France,
3) Laboratory of Gene Technology, Department of Biosystems, KU Leuven, Leuven, Belgium,
4) Institut Pasteur, Laboratoire Pathogenèse des bactéries anaérobies, Département de Microbiologie, Paris, France,
5) Institute of Molecular Systems Biology, Eidgenössische Technische Hochschule (ETH) Zürich, Zürich, Switzerland,
6) Department of Molecular Biosciences, Institute for Cellular and Molecular Biology, Center for Infectious
Disease, University of Texas, Austin, Texas, United States of America
☯Contributions égales de ces auteurs.