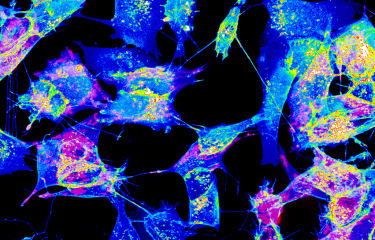
L’organisme humain est constitué de dizaines de milliers de milliards de cellules, toutes issues de la division successive d’une cellule mère en deux cellules filles. Des chercheurs de l’Institut Pasteur ont étudié les midbodies, structures clés dans cette division cellulaire. Ils ont identifié le rôle de BST2, un facteur de restriction impliqué dans les infections virales.
Pour se diviser, les cellules commencent par répliquer à l’identique leurs chromosomes puis les distribuent aux cellules filles. La cellule mère doit ensuite se couper physiquement en deux cellules filles, un processus complexe appelé « cytocinèse ». Des défauts de cytocinèse résultent en la formation de cellules génétiquement instables et qui seraient à l’origine de plus de 40% des cancers humains.
Les « midbodies », structures clés dans la cytocinèse
La cytocinèse commence par la contraction de la cellule mère en son milieu, aboutissant à deux cellules filles reliées entre elles par un pont intercellulaire. Au centre de ce pont, se trouve une structure particulière, le « midbody » qui concentre l’ensemble des protéines nécessaires à la coupure finale des deux cellules filles. Suite à l’abscission, qui a en fait lieu de part et d’autre du midbody, un « midbody remnant » (MBR) est ainsi libéré à l’extérieur des cellules. Ce MBR est actuellement l’objet d’intenses recherches, car il contiendrait des informations influençant la prolifération et la différentiation des cellules qui le captureraient. Cependant, on connait mal les mécanismes permettant aux MBR d’être capturé ou retenu par les cellules cibles.
Découverte de l’implication de la protéine BST2 dans les midbodies
Dans le département Biologie Cellulaire et Infection, l’Unité Trafic Membranaire et Division Cellulaire (Institut Pasteur - CNRS, dirigée par Arnaud Echard) s’intéresse aux mécanismes moléculaires associés à la cytocinèse dans les cellules humaines. Récemment, cette équipe a dressé l’inventaire des protéines qui se concentrent au niveau du midbody avant l’abscission . De façon inattendue, la protéine Tetherin/BST2 y a été identifiée. BST2 est bien connue des virologistes comme un facteur de restriction qui retient à la surface des cellules infectées de nombreux virus enveloppés (notamment le retrovirus HIV-1 responsable du SIDA), limitant ainsi leur dissémination dans l’organisme.

Midbody dans un pont (en rouge), BST2 en cyan pendant la cytocinèse, avant l’abscission. Crédit : Adrien Presle, Audrey Salles, Arnaud Echard/ Institut Pasteur
Un parallèle entre le rôle de BST2 dans les infections virales et dans la capture des midbodies
Des chercheurs dans cette Unité de recherche montrent maintenant que, de façon analogue, cette même protéine contribue à retenir et à capturer les MBRs générés au cours des division cellulaires. Comme dans le cas des virus, l’absence de BST2 favorise la libération des MBR dans le milieu extérieur, leur dissémination à longue distance et réduit leur capture par les cellules cibles.
Ces travaux révèlent ainsi une fonction clé de BST2 dans les cellules non infectées. Ils montrent également un parallèle surprenant entre les virus et une structure naturellement générée au cours des divisions cellulaires. La protéine BST2 est fréquemment sur-exprimée dans les tumeurs humaines, et ceci pourrait potentiellement favoriser la capture des MBRs et par conséquent contribuer à la prolifération cellulaire.
Source:
The Viral Restriction Factor Tetherin / BST2 Tethers Cytokinetic Midbody Remnants to the Cell Surface, Current Biology, 11 mars 2021
Adrien Presle1,2, Stéphane Frémont1, Audrey Salles3, Pierre-Henri Commere4, Nathalie Sassoon1, Clarisse Berlioz-Torrent5, Neetu Gupta-Rossi1,6 and Arnaud Echard1,6
1 Traffic and Cell Division Lab, Institut Pasteur, UMR3691, CNRS, 25-28 rue du Dr Roux, F-75015 Paris, France
2 Sorbonne Université, Collège doctoral, F-75005 Paris, France
3 UTechS Photonic BioImaging PBI (Imagopole), Centre de Recherche et de Ressources Technologiques C2RT, Institut Pasteur, F-75015 Paris, France
4 UTechS CB, Centre de Recherche et de Ressources Technologiques C2RT, Institut Pasteur, F-75015 Paris, France
5 Université de Paris, Institut Cochin, INSERM, CNRS, F-75014 Paris, France
6These authors contributed equally