Résistances, résiliences. Face aux changements, s'adapter, c'est durer.

L’adaptation est un phénomène universel. Résistances microbiennes, inventivité de la réponse scientifique, évolution de notre système immunitaire, l’adaptation est le caractère commun à toutes les choses qui dépassent leurs limitations. Les processus d’adaptation dévoilent ici toute leur complexité dans ce dossier thématique.
Sommaire
Avant-propos - L’adaptation : le secret pour survivre
Définition - Qu'est-ce que l'adaptation ?
Grand format - Chroniques de la résistance
Focus - S'adapter pour mieux piquer
Chronologie - Quand notre système immunitaire s'adapte
Fiche maladie - Hépatite B : adapter la détection et la prise en charge
L’adaptation : le secret pour survivre
Avec Erik Orsenna, romancier et académicien français, ambassadeur de l’Institut Pasteur

Huit millions d’espèces d’insectes et seulement huit mille chez nous, les vertébrés...Comment expliquer leur formidable succès ? Chaque fois que je m’interroge sur la vie, je questionne le professeur Gilles Bœuf*.
Le succès des insectes, j’y vois six raisons, que l’on peut résumer en une équation : Petitesse + manger de tout + habiter partout + se reproduire frénétiquement + aimer la société + diversité = génie de l’adaptation.
Voilà le secret pour survivre : l’adaptation !
D’ailleurs, les insectes ne sont pas seuls à vivre de cette agilité. Les êtres vivants, les populations s’adaptent en permanence. Et bien sûr les pathogènes, forcément les plus filous.
Les mots eux aussi se déguisent, se marient et vivent des transformations constantes. Et, si les langues s’adaptent ainsi de génération en génération, c’est parce qu’elles aussi sont vivantes !
*NDLR : biologiste, ancien président du Muséum national d’histoire naturelle à Paris.
Qu'est-ce que l'adaptation ?
Avec Simonetta Gribaldo, responsable de l’unité Biologue évolutive de la cellule microbienne
Stade 1
Adaptation, n.f : action d’adapter ou de s’adapter à quelque chose (Larousse). L’adaptation est une réaction, une poussée de l’ordre de la nécessité. On (s’) adapte pour convenir, pour s’ajuster à une situation ou à son environnement.
Stade 2
Chez les êtres vivants, l'adaptation est également un processus biologique nécessaire, un ajustement bénéfique, une façon de profiter pleinement de son environnement, d'en tirer le meilleur. L'adaptation relève de la capacité à se réinventer. Elle est un chemin itératif qui bouscule en permanence le statu quo dans un monde en constant changement. C’est un rapport de l’un au tout qui l’entoure, le conditionne et qu’il modifie en retour.
Quant aux scientifiques, ils ne font pas exception ! Les chercheuses et les chercheurs font aussi preuve d’adaptation. La démarche scientifique est une adaptation permanente : de nos connaissances, de nos hypothèses, de nos procédés, de notre conception du monde. L’adaptation nous met face à l’impermanence et nous pousse à l’agilité, à l’humilité et à l’inventivité. Car si elle est nécessaire, au sens inévitable, elle est surtout motrice, animée par l’interdépendance qui caractérise notre existence.

Culture de différents champignons dans des boites de Petri - Crédit : Institut Pasteur - photo François Gardy
Chroniques de la résistance
Avec Paola B. Arimondo, responsable de l’unité Chimie biologique épigénétique, et Philippe Glaser, responsable de l’unité Écologie et évolution de la résistance aux antibiotiques.
Microbes et antimicrobiens : une lutte sans fin
C’est un phénomène à l’ampleur spectaculaire qui inquiète aux quatre coins du globe : la résistance aux agents antimicrobiens a été considérée en 2019 comme l’une des dix plus grandes menaces pour la santé publique mondiale par l’OMS. Les bactéries résistantes aux antibiotiques ont été, à elles seules, responsables de 1,3 million de morts en 2019. L’Institut Pasteur en a fait l’un des trois axes prioritaires de son plan stratégique la même année. Si le haut de l’affiche est occupé par la résistance des bactéries aux antibiotiques, le problème est bien plus large. Antiviraux, antifongiques, antiparasitaires, toutes les catégories de médicaments utilisés pour prévenir et traiter les infections chez l’être humain, l’animal et même le végétal rencontrent de plus en plus de résistances.
La situation est aussi alarmante que compréhensible : qu’un micro-organisme évolue au fil du temps pour contrer les stratégies destinées à l’éradiquer est un processus naturel. Mais l’échappement thérapeutique qui en résulte met à mal notre capacité à contenir des infections sur lesquelles nous avions temporairement pris le dessus. Propagation des épidémies, résurgence de maladies infectieuses que l’on pensait disparues, prise en charge des patients de plus en plus difficile, coûts croissants pour les systèmes de santé : les conséquences de ces résistances sont nombreuses.
En Afrique subsaharienne, 50% des nourrissons positifs au VIH sont infectés par un virus résistant aux antiviraux habituels. Après l’Asie du Sud-Est et le Pacifique occidental, l’Institut Pasteur a mis au jour un nouveau foyer de résistance du parasite du paludisme à l’artémisinine (le dernier antipaludéen mis sur le marché) en Érythrée. Ailleurs, ce sont les moustiques qui déjouent les insecticides, ou les champignons comme Candida auris, bien connu des hôpitaux, qui ne répondent plus aux antifongiques communs. Partout où cette résistance se développe, la communauté scientifique tente de comprendre, prévenir et innover pour inventer de nouvelles stratégies de réponse dans cette lutte sans fin.
Pourquoi et comment les bactéries résistent-elles ?
Depuis la découverte de la pénicilline par Sir Alexander Fleming en 1928, les antibiotiques constituent l’une des plus importantes avancées de la médecine moderne. Ces médicaments, qui peuvent être à spectre large ou à spectre ciblé, permettent de tuer des bactéries ou de bloquer leur prolifération. Mais les bactéries fonctionnent comme tout organisme vivant : face à une menace pour leur survie, elles évoluent. De la même manière que la sélection naturelle a permis aux animaux et donc à l’humain et au système immunitaire de s’adapter à son environnement pathogène, les bactéries ont développé des résistances à nos tentatives de les éradiquer ou au minimum de les contenir. C’est l’antibiorésistance.
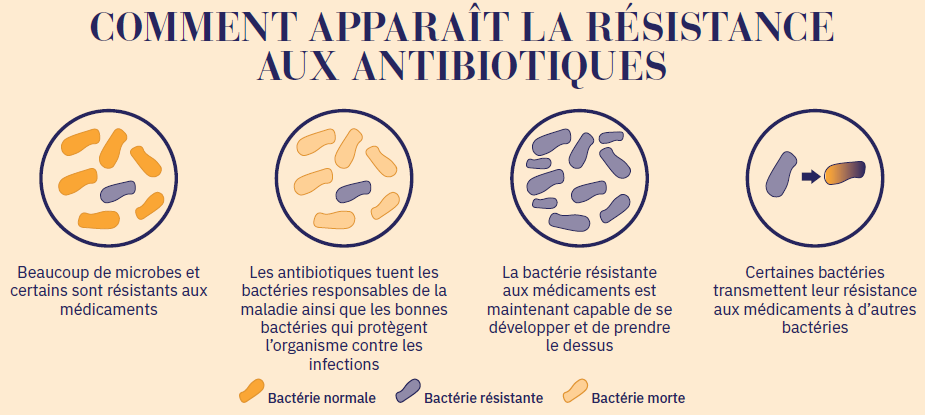
Une bactérie peut acquérir cette qualité de deux manières. Tout d’abord par mutation. Dans ce cas, la bactérie va développer par elle-même une insensibilité à un antibiotique et, grâce à la sélection naturelle, se reproduire mieux et donc devenir majoritaire. C’est par exemple le seul mécanisme mis en œuvre par Mycobacterium tuberculosis, l’agent de la tuberculose humaine et le mécanisme principal pour Pseudomonas aeruginosa, bactérie infectant les poumons des personnes atteintes de mucoviscidose. La résistance aux fluoroquinolones chez Escherichia coli est également essentiellement due à des mutations. Autre possibilité, l’acquisition de gènes de résistance. Les bactéries s’échangent du matériel génétique, les plasmides, qui porte la capacité à résister. Ces transmissions peuvent s’opérer entre bactéries de genres très différents, et entre bactéries pathogènes et non-pathogènes. C’est le mécanisme principal pour Escherichia coli ou pour les staphylocoques dorés résistants à la méticilline par exemple.
Ces réservoirs de résistance aux antibiotiques, portés notamment par les bactéries commensales de nos microbiotes et qui sont fréquemment bénéfiques, sont alimentés par notre utilisation des antibiotiques, notamment ceux à spectre large qui attaquent sans discriminer. Plus on élimine les bactéries non-résistantes, plus on fait de la place à celles qui ont survécu (les résistantes). Mauvais dosage, utilisation en cas d’infection virale (donc non nécessaire), non-respect de la durée du traitement, recours à des produits périmés ou frauduleux, les antibiotiques sont une arme à double tranchant, qui doivent être employés avec pertinence, de façon ciblée, si nous ne voulons pas perdre un terrain difficilement gagné.
Les bactéries résistantes sont à l’origine d’infections plus difficiles voire impossibles à traiter. Certaines peuvent être résistantes à plusieurs antibiotiques, on parle de bactéries multirésistantes (ou BMR). Elles peuvent même être résistantes à tous les antibiotiques existants (panrésistantes) et mener à des impasses thérapeutiques. Parmi les BMR les plus inquiétantes, on retrouve les entérobactéries Escherichia coli et Klebsiella pneumoniae (bactéries du tube digestif responsables d'un très grand nombre d'infections) ; Mycobacterium tuberculosis ; les staphylocoques dorés (Staphylococcus aureus) ou Pseudomonas aeruginosa, fréquemment responsables d’infections nosocomiales. Certaines bactéries résistantes peuvent être à l’origine d’épidémies de grande ampleur, comme au Yémen qui fait face à la plus sévère épidémie de choléra de notre époque et pour laquelle l’Institut Pasteur a pu identifier le plasmide porteur de la résistance à de multiples antibiotiques de la bactérie responsable : Vibrio cholerae. A cela s’ajoute la résistance aux antiparasitaires, antiviraux et antifongiques (comme cité plus haut).
Ennemies publiques
Les sept bactéries les plus traquées pour leur antibiorésistance à l’hôpital :
- Enterococcus faecium, bactérie intestinale ayant développé une résistance à la vancomycine
- Staphylococcus aureus, présente dans les voies pulmonaires et sur la peau en temps normal, de plus en plus résistante à la méticilline et plus rarement à la vancomycine
- Klebsiella pneumoniae, naturellement présente dans le sol, et le microbiome de la peau, de la bouche et des intestins, a développé une résistance aux antibiotiques de la famille des carbapénèmes
- Acinetobacter baumannii, source de beaucoup d’infections nosocomiales, résistante à de nombreux antibiotiques, notamment à tous ceux de la classe des bêta-lactamines
- Pseudomonas aeruginosa, vit dans les sols, les milieux humides et environnements extrêmes, très difficile à traiter car résistante à de nombreux antiseptiques et antibiotiques
- Enterobacter spp., cause de nombreuses infections urinaires et pulmonaires mais encore sensible à la colistine et à la tygécycline
- Escherichia coli, bactérie intestinale souvent inoffensive voire bénéfique mais dont certaines souches sont pathogènes et multirésistantes
La contre-attaque
La résistance aux antimicrobiens est un phénomène global qui affecte tout le vivant. Une surveillance mondiale est en place, lancée en 2015 par l’OMS et son système de surveillance GLASS (Global Antimicrobial Resistance and Use Surveillance System), et de nombreuses bases de données ont été créées pour répertorier chaque apparition d’antibiorésistance dans le monde. Depuis 2002, l'Union Européenne a interdit l’ensemble des antibiotiques utilisés comme facteurs de croissance pour l’élevage des animaux. Une mesure qui s’inscrit dans l’approche scientifique One Health (Une seule Santé), approche qui s’est progressivement développée depuis le début du XXIe siècle. et qui envisage la santé sous le prisme humain-animal-environnement. En France, depuis 2022, l’antibiorésistance fait l’objet d’un plan national porté par le ministère du Travail, de la Santé et des Solidarités.Mais, si cette prise de conscience est globale, les disparités de pays à pays mettent en relief des situations très hétérogènes et révèlent non pas une mais bien des antibiorésistances. Une équipe pluridisciplinaire de l’Institut Pasteur a développé un modèle statistique permettant d’analyser les données collectées depuis 2004 dans 51 pays. Leur conclusion : les déterminants conduisant à l’antibiorésistance sont nombreux, allant des facteurs socio-économiques (PIB par habitant) et climatiques (exposition à des évènements météorologiques extrêmes, température moyenne du pays) à la qualité du système de santé, en passant par le volume d’antibiotiques consommés. Les approches de prévention de la dissémination de la résistance aux antimicrobiens doivent être adaptées au contexte local et aux agents infectieux qui sont ciblés.
Pour autant, la contre-attaque s’organise autour de quelques principes immuables. D’abord mieux comprendre l’émergence et la diffusion de la résistance en alliant notamment épidémiologie, génomique, statistique, biologie évolutive, biologie structurale, chimie biologique et modélisation. Ces recherches permettent de calibrer au mieux l’utilisation des antimicrobiens existants et de rationaliser leur utilisation. Ensuite, identifier de nouveaux médicaments et stratégies thérapeutiques en réponse à la résistance : synthèse de molécules chimiques spécialisées, exploration de nouvelles cibles (comme celles impliquées dans l’interaction hôte-microbes et dans le cycle de vie des agents infectieux), réalisation de criblages phénotypiques et moléculaires, phagothérapie (découverte à l’Institut Pasteur en 1917). La prévention (vaccins, mesures de santé, formation et éducation), la surveillance et le développement de diagnostics sont également fondamentaux dans la lutte contre la résistance aux antimicrobiens. Toutes les pistes doivent être explorées. Le recours à l’intelligence artificielle pour traiter de grandes quantités de données, aider à la découverte de nouvelles molécules et découvrir des biomarqueurs est particulièrement prometteur. Enfin, mieux prévenir en encadrant davantage l’usage des antimicrobiens partout dans le monde et dans toutes les dimensions de la santé humaine et animale. Il est également essentiel d’améliorer le traitement des eaux usées ou de limiter les interactions humain-animal. L’objectif est clair : repousser autant que possible l’avènement d’une ère post-antibiotiques où des infections courantes redeviendraient mortelles.
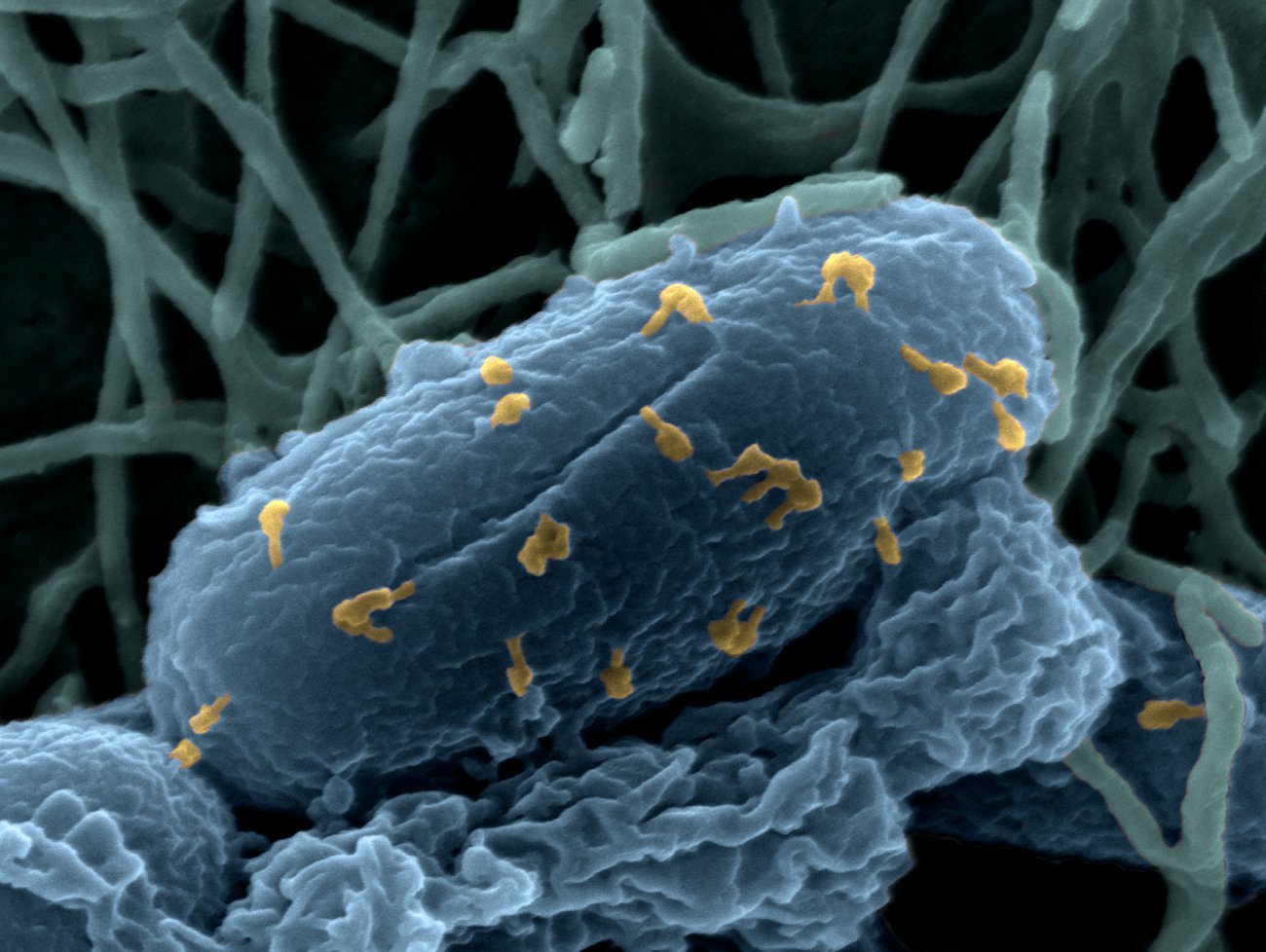
Bactériophages sur la bactérie Escherichia coli - Crédit : Institut Pasteur/Chantal Le Bouguénec, Laurent Debarbieux, Perrine Bomme, Jean-Marc Panaud.
Arsenal
Quelques-uns des fronts de recherche de l’Institut Pasteur en matière de lutte contre l’antibiorésistance :
- Nouvelles thérapies et phagothérapie
Des scientifiques de l’Institut Pasteur et du CNRS ont démontré in vivo que les bactéries sont capables de réguler l’expression de leurs gènes pour échapper aux nombreux bactériophages présents dans l’environnement intestinal (Cell Host & Microbe, 13 avril 2022). La même équipe a mis au point un modèle mathématique pour prédire l’efficacité de la phagothérapie face aux bactéries pathogènes dans le traitement des infections pulmonaires (Cell Reports, 17 mai 2022).
- Compréhension de la résistance aux antibiotiques
Les plasmides sont des molécules d’ADN indépendante du chromosome qui peuvent porter des gènes de résistance aux antibiotiques. Ils sont susceptibles d’être transmis d’une bactérie à une autre par un processus nommé conjugaison bactérienne. Des scientifiques de l’unité Génomique évolutive des microbes ont mis en évidence que certaines de ces molécules contiennent des séquences leur permettant de détourner des protéines issues d’autres plasmides afin d’être transférées d’une bactérie à une autre : ce qu’on peut appeler une conjugaison par auto-stop. Grâce à leurs travaux, il est désormais possible de prévoir les mécanismes de transfert de 90% des plasmides connus chez les bactéries (Nucleic Acids Research, 29 novembre 2022).
Une autre équipe, composée de scientifiques de l’unité Génétique des biofilms et de l’Université Rakuno Gakuen d’Hokkaido au Japon, a réussi à démontrer que les biofilms formés par les bactéries pour survivre dans leur environnement favorisent l’émergence d’antibiorésistances (Communications Biology, 16 mars 2023).
S'adapter pour mieux piquer
Avec Anna-Bella Failloux, responsable de l’unité Arbovirus et insectes vecteurs
Face aux moustiques, vecteurs de certaines des maladies épidémiques à la progression la plus inquiétante, les stratégies de lutte antivectorielle tentent de garder un temps d’avance. Mais les adaptations et les résistances déployées par les moustiques du genre Aedes ou les pathogènes transmis par ces insectes d’autres genres remettent régulièrement en cause les protocoles de prévention, de dépistage et de traitement. Deux études publiées par des équipes de l’Institut Pasteur en 2023 lèvent le voile sur ces évolutions.
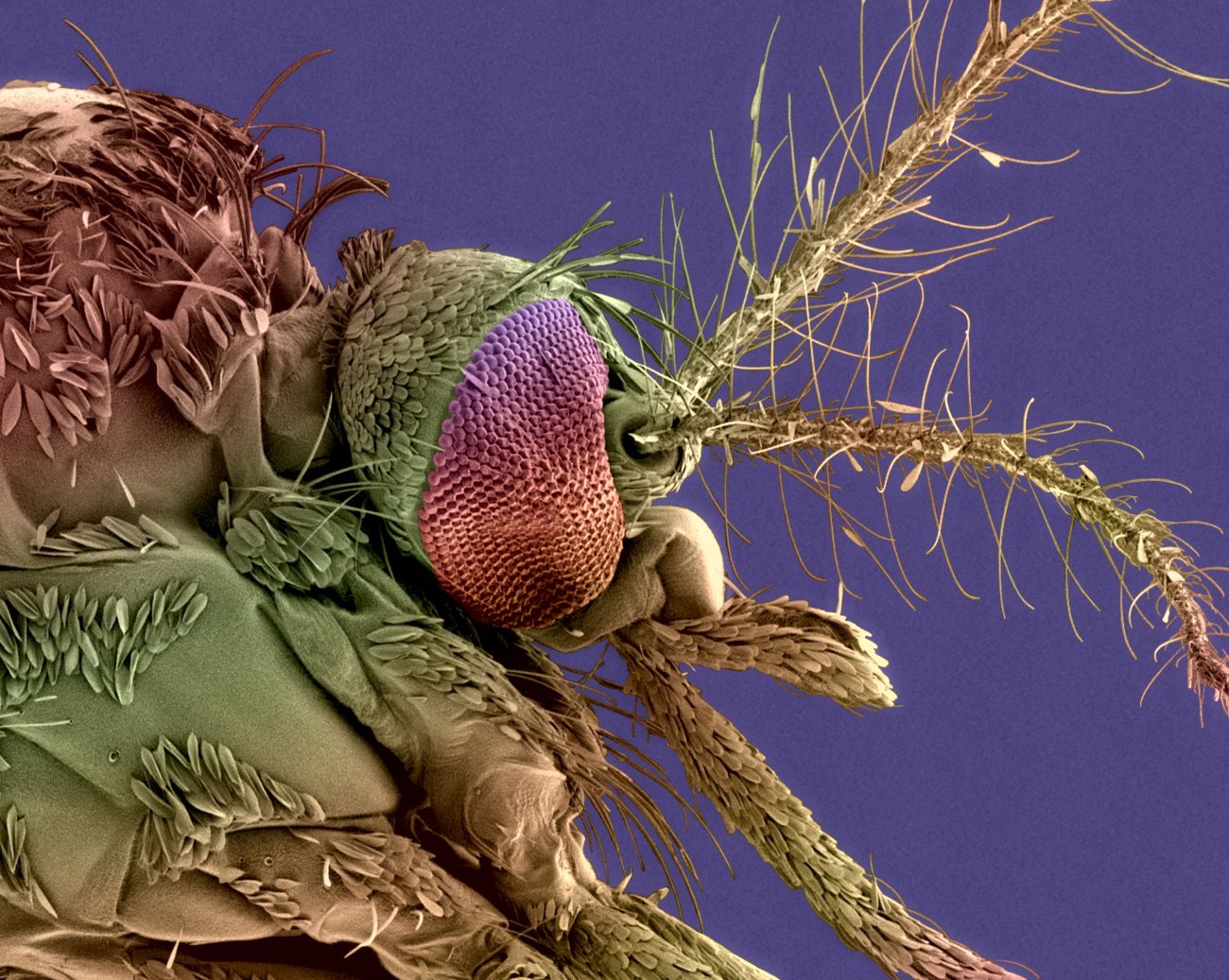
Tête de moustique femelle Aedes albopictus, vecteur du virus de la dengue et du chikungunya. Microphotographie électronique à balayage, image colorisée. - Crédit : Institut Pasteur/Christine Schmitt, Plate-Forme Microscopie Ultrastructurale - Anubis Vega Rua, Laboratoire Arbovirus et Insectes Vecteurs - Colorisation Jean-Marc Panaud.
Le moustique tigre prend ses quartiers en zone tempérée
Le moustique tigre (Aedes albopictus) a bien voyagé depuis sa terre natale d’Asie du Sud-Est. Originaire des zones tropicales, on le trouve aujourd’hui en masse dans les régions tempérées, de l’Amérique à l’Europe, dont la France, où il a déjà envahi 4/5e de l’hexagone. Mais Aedes albopictus ne vient pas seul : c’est l’un des deux vecteurs du virus chikungunya, il est à l’origine des cas autochtones déclarés loin des zones endémiques depuis 2007, en Europe notamment.
Une équipe de scientifiques de l’Institut Pasteur et de l’Université Paris Cité s’est intéressée à la question des effets du climat, en particulier de la température, sur la transmission des pathogènes par les moustiques. Habitué à des moyennes de 28°C, le moustique tigre semble en effet ne pas être empêché par des températures plus basses pour disséminer le virus.
Les résultats de l’étude publiée dans le Journal of Travel Medicine révèlent un exemple d’ajustement mutuel entre le virus et son vecteur en réponse à l’environnement. La température modifie profondément l’expression des gènes et le microbiome bactérien du moustique, ainsi que la diversité génétique du virus du chikungunya. Malgré des processus moléculaires très distincts, le moustique est capable de transmettre le virus avec la même efficacité à 20°C et à 28°C. Une adaptation qui fait craindre l’expansion du chikungunya en l’absence de vaccins et de traitements.
Le paludisme sous le radar en Érythrée
Plasmodium falciparum, parasite responsable des formes les plus graves du paludisme, est depuis quelques années de plus en plus résistant aux techniques de dépistage rapide et de traitement reposant sur l’artémisinine. Une étude coordonnée par des scientifiques de l’Institut Pasteur a mis à jour un nouveau foyer de résistance en Érythrée. C’est le plus inquiétant à date car il révèle la capacité de ces parasites à échapper aux tests rapides, et celle de leurs vecteurs à résister aux insecticides. Les conclusions de ces travaux plaident pour la mise en place urgente de nouvelles stratégies de lutte antivectorielle pour contrer ces adaptations.
Quand notre système immunitaire s'adapte
Avec Lluis Quintana-Murci, responsable de l’unité Génétique évolutive humaine
Dans les années 1950, le chercheur J.B.S. Haldane émet l’hypothèse que la mutation génétique responsable des anomalies des globules rouges observée chez certaines populations africaines se maintient dans la population par la protection qu’elle confère contre le paludisme. L’hypothèse considérant les agents pathogènes comme une des pressions de sélection les plus fortes auxquelles l’être humain a été confronté a depuis été confirmée par de nombreuses études. Mais quels sont les impacts de cette pression de sélection sur notre système immunitaire ? Et avec quelles conséquences sur le risque de développer des maladies inflammatoires ou auto-immunes ? Une étude pilotée par une équipe de l’Institut Pasteur a permis d’analyser les mutations génétiques sur plusieurs centaines d’individus ayant vécu en Europe ces 10000 dernières années, permettant ainsi de retracer l’évolution du système immunitaire humain.
C'est quoi la paléogénomique ?
La paléogénomique est une discipline relativement récente qui étudie l’ADN provenant de restes fossiles humains, animaux, végétaux ou même microbiens. Elle a déjà permis d’importantes découvertes concernant l’histoire et l’évolution de l’être humain et de ses maladies, et a même débouché sur un prix Nobel de médecine, décerné en 2022 à Svante Pääbo pour le séquençage du génome de Néandertal. À l’Institut Pasteur, la paléogénomique a notamment été utilisée pour reconstituer l’histoire de la tuberculose et en comprendre les impacts sur le génome humain.
Un vieux mystère enfin élucidé
Les scientifiques de l’Institut Pasteur, de l’Université Paris Cité, du CNRS et du Collège de France ont utilisé la paléogénomique pour remonter 10 000 ans de notre histoire et tenter de tracer l’évolution dans le temps de centaines de milliers de mutations génétiques. Cette étude, publiée dans la revue Cell Genomics, repose sur l’analyse de la variabilité des génomes de plus de 2 800 individus ayant vécu en Europe sur une période couvrant le néolithique, l'âge du bronze, l'âge du fer, le Moyen Âge et le présent.
Ce travail de détectives du temps a permis de mettre en lumière un ensemble de mutations intervenues en réponse à une sélection naturelle « positive », réduisant le risque de développer des maladies infectieuses. Ces évènements, témoignant d’une adaptation génétique à l’environnement pathogénique, ont débuté relativement récemment sur l’échelle de l’histoire de l’Humanité, à l’âge du bronze, soit il y a environ 4 500 ans. Cette accélération « soudaine » de l’adaptation s’explique par l’intensification de la croissance démographique à cette période et par la propagation des maladies infectieuses du fait de l’intensification des échanges entre populations. En parallèle, les scientifiques ont également pu constater que les mutations associées au risque accru de troubles inflammatoires – dont la maladie de Crohn – ont, elles, augmenté en fréquence au cours des 10 000 dernières années.
« Ces résultats suggèrent que les deux phénomènes sont liés : le risque de troubles inflammatoires ou auto-immuns a augmenté, en tout cas en grande partie, en raison d'une sélection positive de mutations permettant de mieux résister aux maladies infectieuses », explique Lluis Quintana-Murci.
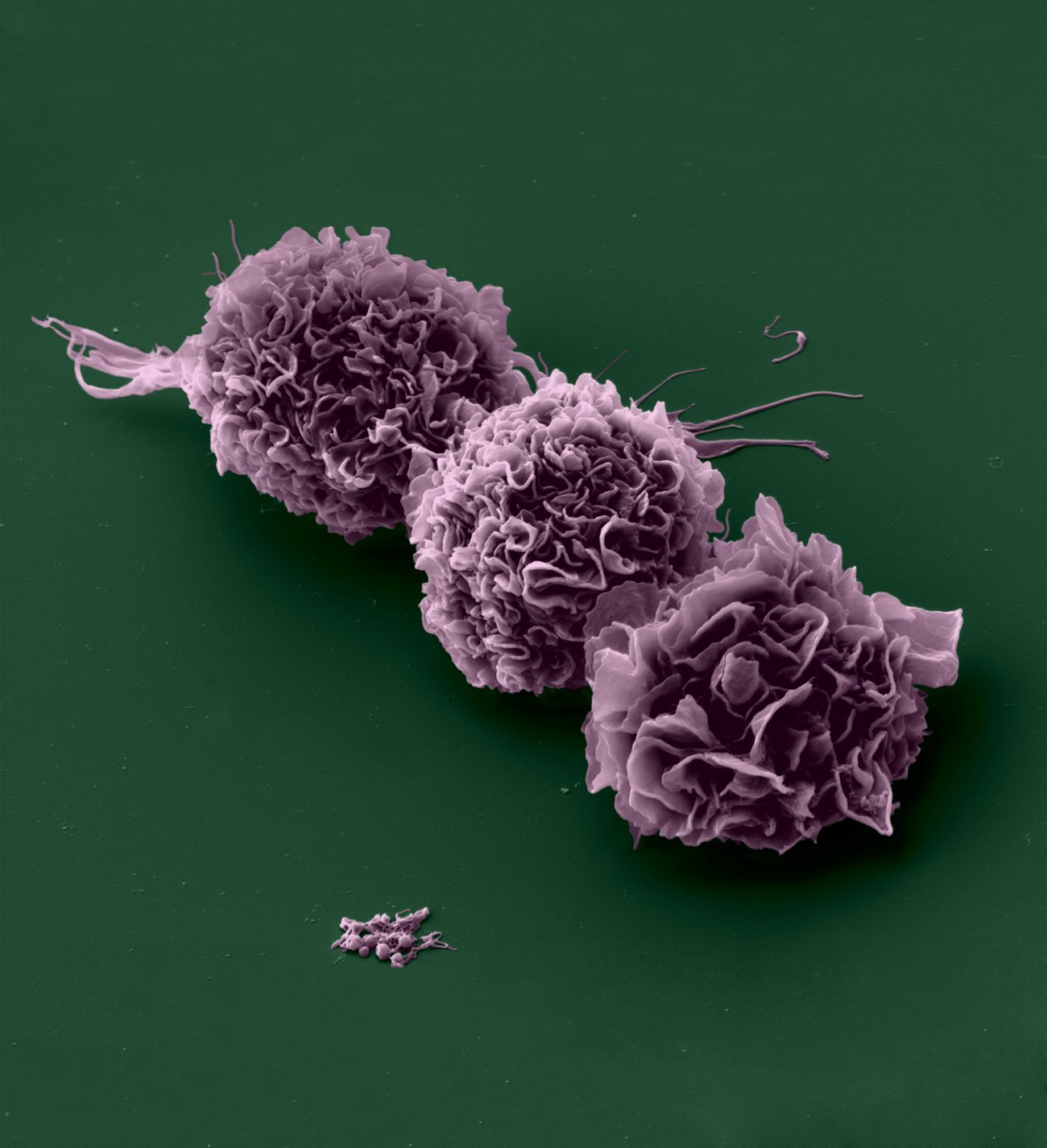
Cellules dendritiques - Crédit : Institut Pasteur/Olivier Schwartz, Plateforme de Microscope électronique. Colorisation Jean Marc Panaud
Hépatite B : Adapter la détection et la prise en charge
Avec Yusuke Shimakawa, chercheur de l’unité Épidémiologie des maladies émergentes
Les hépatites sont des inflammations du foie causées par des virus, plus rarement par des substances toxiques. Ces infections virales sont le fait de cinq pathogènes identifiés par des lettres de A à E. L’hépatite B est l’une des principales maladies affectant l’être humain. Elle a déjà touché plus de deux milliards d’individus. En Afrique sub-saharienne, zone de forte endémie, les scientifiques de l’Institut Pasteur font évoluer les modèles de dépistage pour optimiser la prise en charge des personnes infectées et limiter la propagation de la maladie.
Origines
La première épidémie potentielle enregistrée remonte à 1883, dans un chantier naval de Brême où les ouvriers avaient été vaccinés contre la variole avec la lymphe d’autres personnes, porteuses d’un virus de l’hépatite B encore non identifié. L’antigène à l’origine de l’hépatite B est identifié en 1964 par Baruch Blumberg, scientifique américain et prix Nobel de médecine en 1976 pour sa découverte.
Symptômes, transmission et traitement
Le virus se transmet par tous les liquides et sécrétions biologiques. Les principales voies de transmission sont celle de la mère à l’enfant pendant l’accouchement, le contact étroit avec une personne infectée, les contacts sexuels, l’utilisation de seringues contaminées et les transfusions à risque. Extrêmement infectieux, le virus touche plus de 5% de la population générale en Afrique sub-saharienne, en Asie de l’Est et dans le Pacifique. 254 millions de personnes dans le monde vivent avec une infection chronique par le VHB. La maladie est souvent asymptomatique, ou peut déclencher des symptômes similaires à ceux de la grippe ou communs aux inflammations du foie. Il n’existe pas de traitement spécifique pour l’hépatite aiguë ; il faut compter sur le travail de défense mis en œuvre par le système immunitaire. L’infection chronique est, elle, traitée chez les personnes à haut risque de maladies chroniques du foie par des antiviraux spécifiques. Les vaccins, développés depuis le début des années 1980, ont un taux d’efficacité très élevé pour 98% de la population.
Adaptation au contexte sub-saharien
Les pays les plus touchés sont aussi ceux où les moyens de dépistage et de traitement sont les plus limités. Si beaucoup portent le virus sans symptôme, certains finissent par développer des maladies chroniques pouvant mener jusqu’au cancer. Une approche de diagnostic et traitement universel peut sembler attrayante lorsque l’accès aux tests pour déterminer l’éligibilité aux traitements est limité. Cependant, elle peut également être inadaptée pour des raisons de coût et de difficultés à pérenniser l’accès à des traitements à vie. Les équipes de l’Institut Pasteur ont mis au point le premier modèle mathématique pour déterminer l’éligibilité optimale aux traitements antiviraux dans le contexte de la Gambie. Le résultat suggère une stratégie ciblée et simplifiée, utilisant le test diagnostic TREAT-B, qui associe l’antigène « e » de l’hépatite B et l’alanine transaminase. Il permet de réduire les cas de cirrhose et de carcinome hépatocellulaire avec un coût divisé par deux par rapport à une approche de traitement universel.


