Loin d’être passives, les bactéries sont capables d’interagir avec leur environnement proche. Comment ? Par exemple, en sécrétant des molécules hors de leur enveloppe par le biais de machineries protéiques complexes. Des scientifiques lèvent le voile sur les mécanismes de formation d’un de ces systèmes de sécrétion, ouvrant ainsi la voie à de potentielles applications thérapeutiques.
Les bactéries sont des êtres unicellulaires capables d'interagir avec leur milieu extérieur, par exemple en diffusant des molécules en dehors de leur enveloppe. Le canal qui assure cette diffusion est un complexe géant composé de plusieurs protéines, le système de sécrétion. Les molécules sécrétées étant des facteurs clés de la survie, l'adaptation et la pathogénicité des bactéries, les systèmes de sécrétion représentent par conséquent des cibles thérapeutiques de choix.
Si les différents composants de ces complexes protéiques sont aujourd’hui bien identifiés, leurs structures ne sont que partiellement connues. La dynamique de leurs assemblages et son rôle dans la sécrétion moléculaire restent également à découvrir.
Voyage au cœur de la formation du système de sécrétion de type II
Dans une étude récemment publiée, des scientifiques du département de biologie structurale et chimie de l’Institut Pasteur et du CNRS se sont plus particulièrement intéressés au système de sécrétion de type 2 (SST2). Constitué de 12 à 14 protéines, le SST2 est une machinerie complexe enchâssée dans l’enveloppe de certaines bactéries pathogènes. Il joue un rôle essentiel dans la colonisation et la virulence bactérienne, en sécrétant des molécules permettant l’adhésion cellulaire ou des toxines.
Les chercheuses et chercheurs ont plus précisément étudié la plateforme d’assemblage du SST2. Composée des protéines PulL et PulM, cette plateforme est nécessaire à la constitution du système de sécrétion. « En combinant des approches microbiologiques, biochimiques et biophysiques, nous avons pu déterminer le mode d’assemblage et la structure atomique du complexe formé par des domaines spécifiques de ces deux composants », précise Nadia Izadi-Pruneyre, responsable de l’Unité "Systèmes Transmembranaires Bactériens".
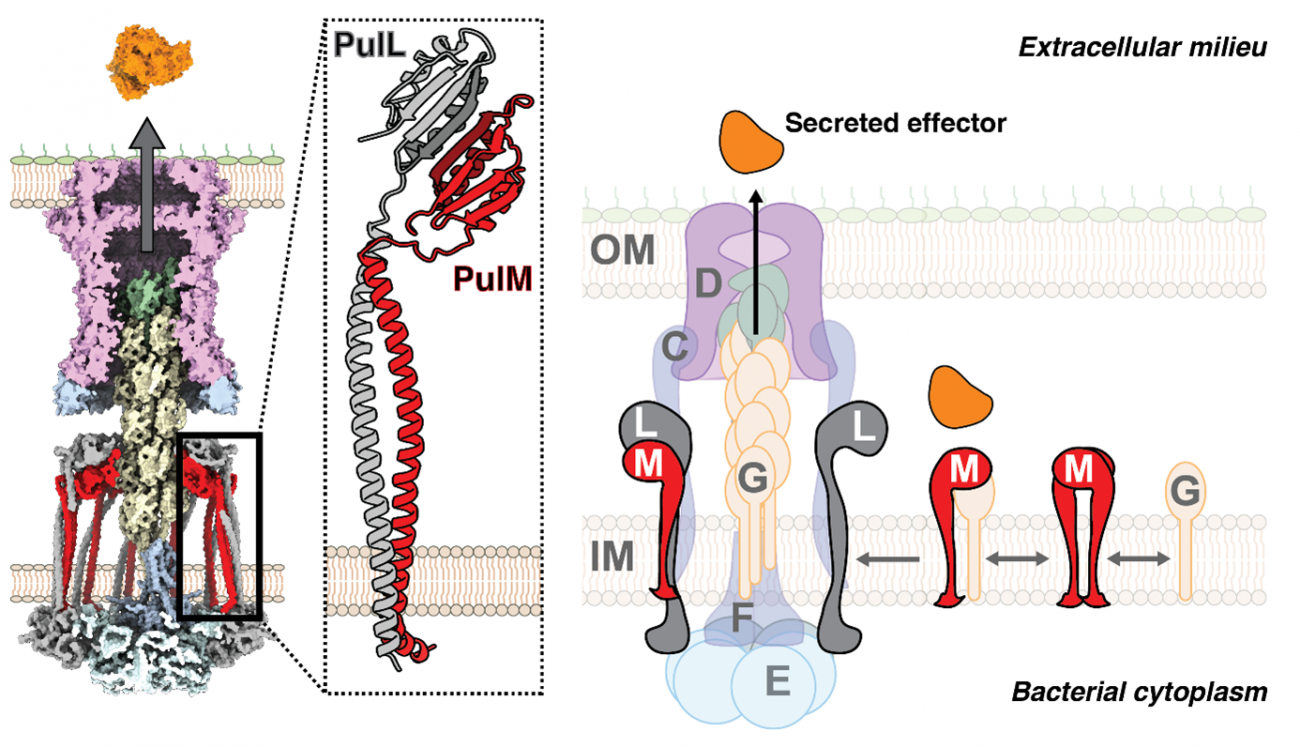
La chercheuse ajoute : « Nous montrons comment la complémentarité structurale et la plasticité de ces structures favorisent leur association au cours du processus de sécrétion. ». Ces résultats suggèrent également que le complexe PulL-PulM orchestre l’assemblage des différents composants du SST2 et joue un rôle clé dans le transport de protéines à l’extérieur de la bactérie.
Cette étude permet une meilleure compréhension de la dynamique moléculaire aboutissant à la formation du SST2. Elle ouvre de nouvelles perspectives de recherche et permet d’envisager l’utilisation de ce système de sécrétion pour de futures applications en thérapie antimicrobienne, en biologie synthétique ou en biotechnologie.
Cette étude entre dans le cadre de l’axe scientifique prioritaire Résistance aux agents antimicrobiens du plan stratégique 2019-2023 de l’Institut Pasteur.
Source :
Structure and dynamic association of an assembly platform subcomplex of the bacterial type II secretion system, Structure, 30 Décembre 2022
DOI : 10.1016/j.str.2022.12.003
Régine Dazzoni1, Yuanyuan Li2, Aracelys López-Castilla1, Sébastien Brier3, Ariel Mechaly4, Florence Cordier1,3, Ahmed Haouz4, Michael Nilges1, Olivera Francetic2, Benjamin Bardiaux1*, Nadia Izadi-Pruneyre1,5,6*
1 Institut Pasteur, Université Paris Cité, CNRS UMR3528, Structural Bioinformatics Unit, 75015 Paris, France
2 Institut Pasteur, Université Paris Cité, CNRS UMR3528, Biochemistry of Macromolecular Interactions Unit, 75015 Paris, France
3 Institut Pasteur, Université Paris Cité, CNRS UMR3528, Biological NMR and HDX-MS Technological Platform, 75015 Paris, France
4 Institut Pasteur, Université Paris Cité, CNRS UMR3528, Crystallography Platform, 75015 Paris, France
5 Institut Pasteur, Université Paris Cité, CNRS UMR3528, Bacterial Transmembrane Systems Unit, 75015 Paris
6Lead contact
*Correspondence: benjamin.bardiaux@pasteur.fr (B.B.), nadia.izadi@pasteur.fr (N.I.-P.)