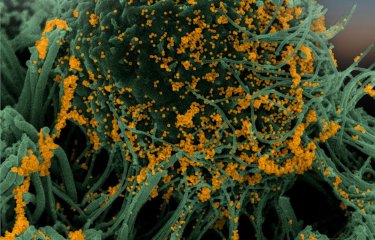
La pandémie de la Covid-19, qui touche le monde depuis plus de deux ans, est causée par le SARS-CoV-2. Le génome de ce virus respiratoire est constitué d’un unique brin d’ARN, une molécule proche de l’ADN qui permet au virus de se répliquer dans l’organisme. Des chercheurs de l’Institut Pasteur, de l’Inserm et de l’APHP ont mis au point une nouvelle méthode, CoronaFISH, pour observer le virus et détecter sa présence.
Depuis le début de la pandémie, un enjeu majeur est d’étudier la biologie du virus dans les cellules et détecter sa présence dans des organes ou patients infectés. Pour cela, il faut des outils permettant de le visualiser par microscopie. « Plusieurs techniques existent actuellement, mais la plupart ont des limitations, soit parce qu’elles sont indirectes, chères ou peu robustes. Par exemple, on peut utiliser des anticorps pour révéler des protéines du virus, mais l’obtention de ces anticorps est longue et coûteuse, leur sensibilité est compromise par les variants et la présence de protéines virales dans une cellule n’implique pas forcément que le virus s’y réplique activement » explique Christophe Zimmer, responsable du laboratoire Imagerie et modélisation à l’Institut Pasteur.
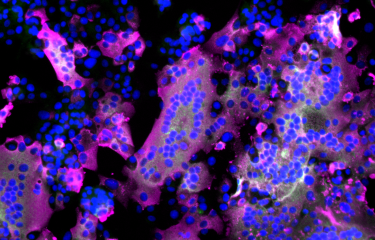
Pour visualiser le génome viral lui-même, des chercheurs de l’Institut Pasteur, de l’Inserm et de l’APHP ont développé une nouvelle méthode. Cette méthode, appelée CoronaFISH, a pour but de marquer l’ARN du virus directement en se basant sur l’hybridation in situ, une technique qui permet de repérer une partie (« séquence ») spécifique de l’ARN en s’accrochant à ses composants, les nucléotides, par complémentarité. En effet, pour chaque séquence il existe une séquence dite complémentaire qui s’accrochera naturellement à la molécule d’ARN. « Cette méthode utilise une batterie de sondes constituées de fragments d’ADN liés à des molécules fluorescentes et complémentaires de la séquence de l’ARN viral » indique Florian Mueller, du même laboratoire, et l'un des auteurs principaux de l'article. « Ainsi, si le virus est présent, les oligonucléotides s’accrochent à l’ARN viral, qui devient donc visible par fluorescence sous un microscope », poursuit-il.
Les scientifiques ont validé CoronaFISH dans des cellules du singe vert africain, un type de cellule couramment utilisées pour l’étude du SARS-CoV-2, ainsi que dans des cellules humaines. « Notre technique a été capable de distinguer des populations cellulaires avec des charges virales différentes, même lorsque seules quelques cellules étaient infectées. Contrairement aux méthodes qui analysent des populations de cellules de façon globale, CoronaFISH permet une analyse spatiale et temporelle de la réplication et de la propagation du SARS-CoV-2 », commente Giovanna Barba-Spaeth, de l'unité de Virologie Structurale, l’une des auteurs seniors de l'article.
De plus, ils ont montré que CoronaFISH peut détecter le virus dans les tissus de poumon d’une personne atteinte du Covid-19, ainsi que dans des échantillons nasopharyngés utilisés pour des tests PCR. Enfin, l’équipe a modifié CoronaFISH pour permettre l’observation de SARS-CoV-2 par microscopie électronique, et donc l’étude du virus dans le contexte de l’ultrastructure cellulaire (structure fine des éléments constituant la cellule).
Cette méthode enrichit la boîte à outils disponible pour l’étude du SARS-CoV-2, car elle permet de visualiser finement l’ARN du virus, en microscopie optique ou électronique, dans les cellules ou les tissus. A ce titre, elle permettra de mieux comprendre quels organes peuvent être atteints par le virus.
Cette méthode pourrait même, une fois optimisée et validée par plus d’études, servir de base à un test diagnostic. Un test basé sur CoronaFISH présenterait des avantages par rapport à la PCR : par exemple, il ne nécessiterait pas d’extraction de l’ARN, et grâce au grand nombre de sondes utilisées, sa sensibilité ne devrait pas souffrir de l’apparition de variants.
Ces travaux ont bénéficié d’un financement AXA COVID-19 project COVID-SPREAD
Le Centre de Recherche Translationnel de l’Institut Pasteur a accompagné le projet pour sa conformité éthico réglementaire. Le projet a également été enregistré sur la plateforme Health Data Hub qui donne un accès au public aux projets de recherche utilisant des données des personnes, données qui peuvent avoir été recueillies à l’origine dans le cadre du soin par exemple.
Cette étude entre dans le cadre de l’axe scientifique prioritaire Maladies infectieuses émergentes du plan stratégique 2019-2023 de l’Institut Pasteur.
Source :
Sensitive visualization of SARS-CoV-2 RNA with CoronaFISH, Life Science Alliance, 7 janvier 2022
Elena Rensen1,Stefano Pietropaoli2, Florian Mueller1, Christian Weber1, Sylvie Souquere3, Sina Sommer2, Pierre Isnard4,5, Marion Rabant4,5, Jean-Baptiste Gibier6, Fabiola Terzi4, Etienne Simon-Loriere7,Marie-Anne Rameix-Welti8,9, Gerard Pierron10, Giovanna Barba-Spaeth2, Christophe Zimmer1
1 Institut Pasteur, Université de Paris, CNRS UMR 3691, Imaging and Modeling Unit, Paris, France
2 Institut Pasteur, Université de Paris, CNRS UMR 3569, Unité de Virologie Structurale, Paris, France 3 Gustave Roussy, AMMICA UMS-3655, Villejuif, France
4 Université de Paris, INSERM U1151, CNRS UMR 8253, Institut Necker Enfants Malades, Département “Croissance et Signalisation”, Paris, France
5 Service d’Anatomo-Pathologie, AP-HP, Hôpital Necker Enfants Malades, AP-HP Centre, Paris, France
6 Service d’Anatomo-Pathologie, Centre de Biologie Pathologie, CHU Lille, Lille, France
7 Institut Pasteur, Université de Paris, G5 Evolutionary Genomics of RNA Viruses, Paris, France
8 Université Paris-Saclay, INSERM, Université de Versailles St. Quentin, UMR 1173 (2I), Montigny-le-Bretonneux, France
9 AP-HP, Université Paris Saclay, Hôpital Ambroise Pare, Laboratoire de Microbiologie, Boulogne-Billancourt, France
10 Gustave Roussy, CNRS UMR 9196, Villejuif, France