Bordetella pertussis est l’agent responsable de la coqueluche, actuellement en recrudescence. La bactérie produit notamment une toxine, CyaA. Un consortium regroupant plusieurs équipes de l’Institut Pasteur, du CNRS et de Sorbonne Université, en étroite collaboration avec les plateformes techniques du Pole Protéine du C2RT et plusieurs lignes de lumières des synchrotrons SOLEIL et ESRF, a étudié le mécanisme d’entrée de la toxine CyaA dans les cellules cibles. Ce travail a mis au jour un mécanisme de translocation membranaire, très intéressant dans la connaissance de la biologie du vivant.
La bactérie Bordetella pertussis est responsable de la coqueluche, une maladie contagieuse, actuellement en recrudescence, qui peut être particulièrement sévère, voire mortelle, pour les nourrissons et les personnes fragiles. Bordetella pertussis produit de nombreux facteurs de virulence, dont la protéine CyaA, une toxine qui contribue aux étapes précoces de colonisation bactérienne des poumons des personnes infectées. Elle intoxique notamment les cellules du système immunitaire inné, en particulier les macrophages alvéolaires qui assurent la défense contre les pathogènes dans les alvéoles de nos poumons.
La translocation membranaire de CyaA, une étape clef du processus d’intoxication cellulaire
La toxine CyaA possède un mécanisme original pour pénétrer à l’intérieur de nos cellules : dans un premier temps, elle se lie à un récepteur spécifique à la surface des cellules cibles, puis une partie de la chaine polypeptidique, le domaine catalytique, est directement transporté à travers la membrane plasmique pour atteindre le cytosol des cellules cibles selon un processus appelé translocation membranaire. Le domaine catalytique de CyaA est ensuite activé par une protéine cellulaire (nommée calmoduline) pour synthétiser de l’AMPc, une molécule essentielle de régulation de la physiologie cellulaire. Cependant, les quantités élevées d’AMPc générées par CyaA entrainent une altération de la physiologie cellulaire, et en particulier l’inhibition des fonctions phagocytaires des cellules du système immunitaire inné, laissant ainsi le champ libre à la colonisation des poumons par la bactérie.
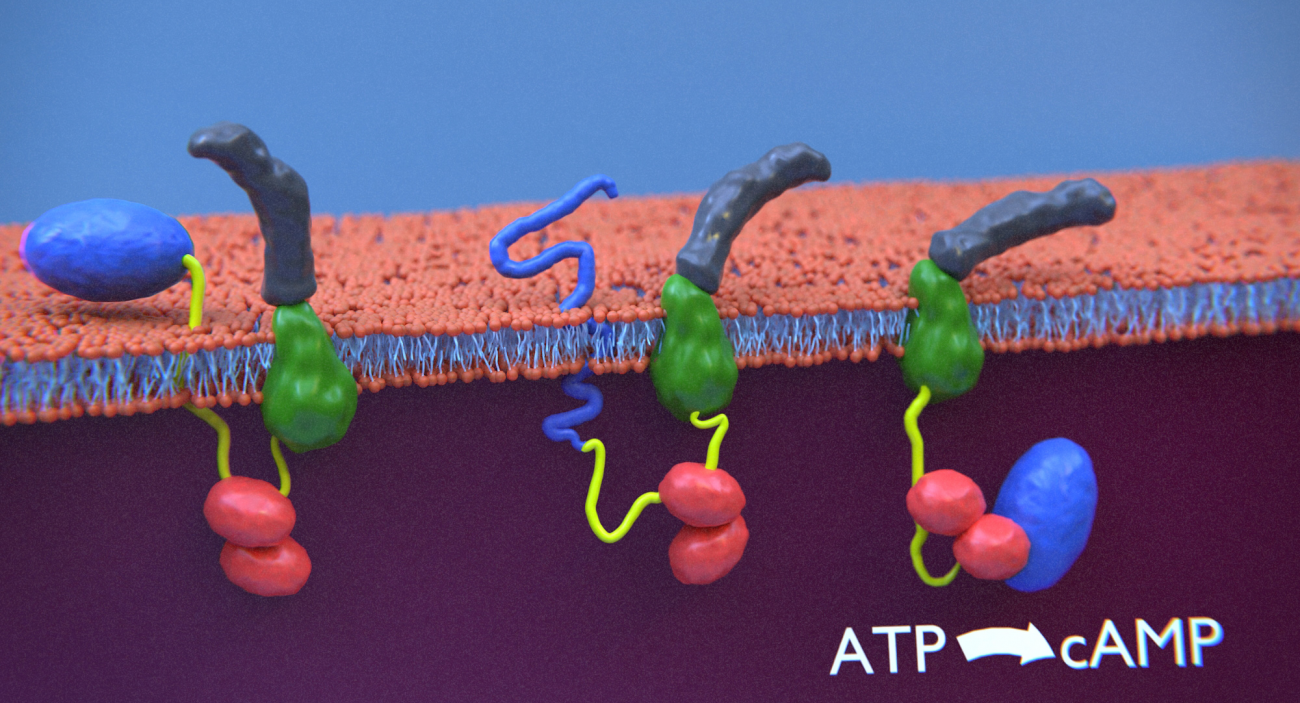
Le mécanisme d'invasion cellulaire par les toxines bactériennes est encore mal compris. La toxine CyaA de Bordetella pertussis, l'agent causal de la coqueluche, transloque directement son domaine catalytique à travers la membrane plasmique. Le segment perméabilisant de la membrane (jaune) de la CyaA se transloque à travers la membrane plasmique et se lie à la calmoduline (rouge), ce qui facilite l'entrée du domaine catalytique (bleu) dans les cellules-hôtes, tandis que les domaines hydrophobe et d'acylation (vert) interagissent avec la membrane et que le domaine C-terminal "Repeat-in-Toxin" (gris) reste dans le milieu extra-cellulaire.
Crédit : Julien Husson https://cellmechanics.jimdofree.com/
La translocation membranaire de CyaA utilise le harponnage moléculaire
Si l’activation du domaine catalytique de CyaA par la calmoduline était connue depuis longtemps, le mécanisme de translocation membranaire restait une énigme. « Nous avons montré qu’une région de CyaA, appelée la région de translocation, ou TR, est capable de traverser la membrane plasmique des cellules cibles de manière similaire aux peptides antibiotiques. Nous avons également montré que cette région TR interagit avec une très forte affinité avec la calmoduline. La région TR « harponne » la calmoduline dans le cytosol : cette interaction piège la région TR dans le cytosol et force le transport du domaine catalytique à travers la membrane », explique Alexandre Chenal, responsable du groupe BiophysiCyaA au sein de l’unité Biochimie des interactions macromoléculaires de l’Institut Pasteur. Ce transport du domaine catalytique, de l’extérieur vers l’intérieur de la cellule, est possible en raison de la forte affinité entre la région TR et la calmoduline. En effet, le domaine catalytique se déplie facilement à la surface de la cellule, ce qui favorise son passage à travers la membrane plasmique, tiré par le complexe formé par la région TR de CyaA et la calmoduline dans le cytosol.
Cette étude permet de mieux comprendre le mécanisme de translocation membranaire des protéines en général et, à l’échelle moléculaire, d’identifier les résidus impliqués dans ce processus. De plus, d’un point de vue thermodynamique, cette étude devrait permettre de quantifier l’énergie (la force de traction en quelque sorte) requise pour transloquer et vectoriser des biomolécules thérapeutiques dans des cellules cibles, comme par exemple des cellules tumorales.
Enfin, cette étude illustre les synergies entre les équipes de recherche, les plateformes du C2RT, notamment la Plateforme de Biophysique Moléculaire, la Plateforme de cristallographie et la Plateforme Technologique de RMN Biologique, ainsi que les lignes DISCO, SWING et PROXIMA (Synchrotron SOLEIL, St Aubin, France) et MASSIF (Synchrotron ESRF, Grenoble, France).
Source :
A High-Affinity Calmodulin-Binding Site in the CyaA Toxin Translocation Domain is Essential for Invasion of Eukaryotic Cells, Advanced Science, First published: 08 March 2021, https://doi.org/10.1002/advs.202003630
Alexis Voegele1,2#, Mirko Sadi1,2#, Darragh P O’Brien1#, Pauline Gehan3#, Dorothée Raoux-Barbot1, Maryline Davi1, Sylviane Hoos4, Sébastien Brûlé4, Bertrand Raynal4, Patrick Weber5, Ariel Mechaly5, Ahmed Haouz5, Nicolas Rodriguez3, Patrice Vachette6, Dominique Durand6, Sébastien Brier7, Daniel Ladant1*, Alexandre Chenal1*
1: Biochemistry of Macromolecular Interactions Unit, Department of Structural Biology and Chemistry, Institut Pasteur, CNRS UMR3528, 75015 Paris, France
2: Université de Paris, Sorbonne Paris Cité, Paris, France
3: Sorbonne Université, École normale supérieure, PSL University, CNRS, Laboratoire des biomolécules, LBM, 75005 Paris, France
4: Plateforme de Biophysique Moléculaire, Institut Pasteur, UMR 3528 CNRS, Paris, France
5: Institut Pasteur, Plateforme de cristallographie-C2RT, UMR-3528 CNRS, Paris, France
6: Université Paris-Saclay, CEA, CNRS, Institute for Integrative Biology of the Cell (I2BC), 91198, Gif-sur-Yvette, France.
7: Biological NMR Technological Plateform, Center for Technological Ressources and Research, Department of Structural Biology and Chemistry, Institut Pasteur, CNRS UMR3528, 75015 Paris, France