Lors d’une infection, la bactérie pathogène et celles de l’intestin interagissent étroitement. Elles peuvent avoir à se partager les ressources, limitant ainsi l’infection du pathogène, ou celui-ci peut tirer profit de cette cohabitation vis-à-vis de sa stratégie infectieuse.
L’infection par Clostridioides difficile est la principale cause des diarrhées infectieuses dans les hôpitaux et les établissements de soins de longue durée. L’exposition aux antibiotiques va, en détruisant le rôle protecteur du microbiote intestinal, favoriser l’émergence de ce bacille pathogène. Ce dernier peut alors se développer sans résistance dans le colon. La manière dont les bactéries commensales, faisant partie du microbiote intestinal, assurent cette protection fait l’objet de nombreux travaux. Une étude internationale publiée dans Cell Host & Microbe, et à laquelle l’Institut Pasteur a participé, vient de définir les mécanismes moléculaires par lesquels le microbiote peut prévenir ou aggraver les infections à C.difficile.
Les bactéries du microbiote entrent en compétition ou favorisent le développement du pathogène
Chez des souris dépourvues de tout germe, les chercheurs ont montré qu’en introduisant des bactéries commensales ayant des capacités métaboliques variées, le microbiote est capable de moduler le pouvoir de virulence de C. difficile et sa survie dans l'hôte. C’est le cas notamment de Paraclostridium bifermentans, une espèce qui concurrence C. difficile pour les nutriments essentiels à sa croissance comme les acides aminés. À l’inverse, Clostridium sardiniense est une espèce capable de nourrir activement C. difficile par les produits de son métabolisme. Ainsi les souris colonisées par P. bifermentans survivent à l'infection à C. difficile tandis que celles colonisées par C. sardiniense succombent rapidement.
Les effets protecteurs ont été reproduits chez des souris conventionnelles infectées recevant P. bifermentans en bactériothérapie orale. « Ces phénotypes de viabilité diamétralement opposés que nous avons observé avec ces deux bactéries commensales, nous ont permis de définir l’impact déterminant des nutriments intestinaux disponibles au cours de la maladie sur les comportements de C. difficile » explique Bruno Dupuy du laboratoire Pathogenèse des bactéries anaérobies de l’Institut Pasteur. Ces mécanismes mettent en évidence des stratégies plus larges par lesquelles le microbiote commensal peut protéger contre les agents pathogènes en limitant les nutriments qui les aident à coloniser et à infecter l'intestin. Ainsi, comme le propose Bruno Dupuy, « en s'appuyant sur l’analyse organisationnelle in vivo des interactions hôte-commensal-pathogène, nous devrions pouvoir développer des bactériothérapies ciblées pour traiter et prévenir ces maladies infectieuses ».
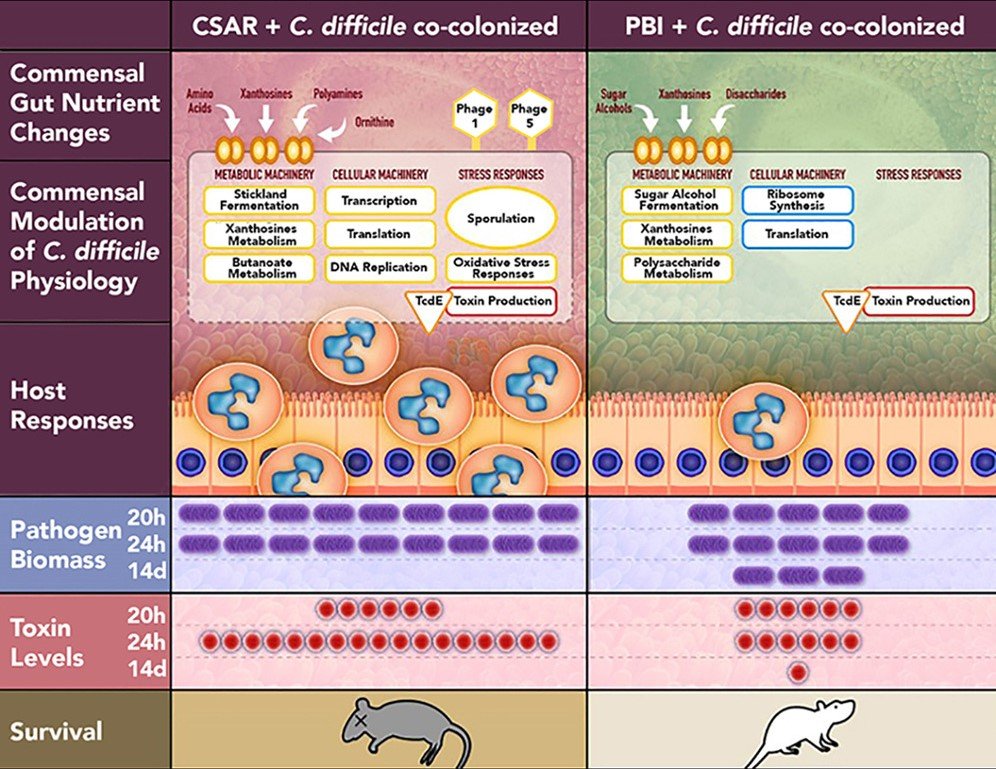
Ce schéma résume le contrôle de la virulence de Clostridioides difficile par des bactéries commensales. Les souris colonisées par Paraclostridium bifermentans (PBI) survivent à l'infection par C. difficile avec une gravité fortement réduite de la maladie, tandis que les souris colonisées par Clostridium sardiniense (CSAR) succombent rapidement. Des analyses systématiques in vivo révèlent comment chaque commensal modifie l'environnement nutritionnel de l'intestin. Ces modifications auront pour effet de moduler le métabolisme, la machinerie cellulaire, les réponses au stress et la production de toxines de C. difficile. Ces changements auront aussi pour conséquence de modifier la réponse de l’hôte qui est un élément important dans la virulence de ce pathogène. Les éléments entourés en jaune et bleu indiquent respectivement une régulation positive et négative des programmes génétiques associés in vivo, l'orange indique des niveaux d'expression constitutifs. © 2019, Massachusetts Host-Microbiome Center
Cette étude entre dans le cadre de l’axe scientifique prioritaire Résistance aux agents antimicrobiens du plan stratégique 2019-2023 de l’Institut Pasteur.
Source :
In vivo commensal control of Clostridioides difficile virulence, Cell Host & Microbe, 11 novembre 2021 https://www.sciencedirect.com/science/article/pii/S1931312821004236
Brintha P. Girinathan1, Nicholas DiBenedetto1, Jay N. Worley1,2, Johann Peltier3,4, Mario Arrieta-Ortiz5, Rupa Immanuel5, Richard Lavin1, Mary L. Delaney1,6, Christopher Cummins1, Maria Hoffman7, Yan Luo7, Narjol Gonzalez Escalona7, Marc Allard7, Andrew B. Onderdonk1,6, Georg K. Gerber1,8, Abraham L. Sonenshein9, Nitin Baliga5, Bruno Dupuy3, Lynn Bry1,7
- Massachusetts Host-Microbiome Center, Dept. Pathology, Brigham & Women’s Hospital, Harvard Medical School, Boston, MA02115, USA
- National Center of Biotechnology Information, National Library of Medicine, Bethesda, MD 20894, USA
- Institut Pasteur, Université de Paris, UMR-CNRS2001, Laboratoire Pathogenèse des Bactéries Anaérobies, F-75015 Paris, France.
- Institute for Integrative Biology of the Cell (I2BC), Université Paris-Saclay, CEA, CNRS, 91198, Gif-sur-Yvette cedex, France.
- Institute of Systems Biology, Seattle, WA 98109, USA
- Clinical Microbiology Laboratory, Department of Pathology, Brigham & Women’s Hospital, Boston, MA.02115, USA
- Center for Food Safety and Applied Nutrition, United States Food and Drug Administration, Department of Health and Human Services, College Park, MD20740, USA
- Harvard-MIT Health Sciences & Technology, Cambridge, MA 02139, USA
- Department of Molecular Biology and Microbiology, Tufts University School of Medicine, Boston, MA 02111, USA