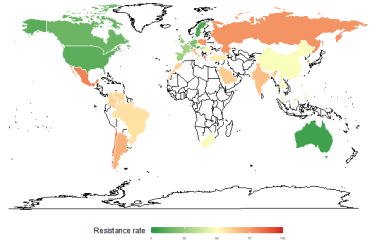
Aujourd’hui, il est difficile de concevoir de nouvelles molécules efficaces pour lutter contre les maladies infectieuses. C’est un immense défi pour les scientifiques face à l’augmentation des résistances bactériennes aux antibiotiques. Des chercheurs de l’unité Plasticité du génome bactérien de l’Institut Pasteur ont relevé ce défi et mis au point un nouvel outil biotechnologique qui contourne les limites d’échantillonnages auxquelles se heurtent les chercheurs et ouvre la possibilité de trouver et d’exploiter de nouvelles molécules performantes.
Les machineries moléculaires qui produisent les antibiotiques sont de deux types : les polycétides synthases et les NRPS (Non-Ribosomal Peptides Synthetase). Depuis les années 40, de nombreuses banques de polycétides synthases et de NRPS ont été passées au peigne fin par l’industrie pharmaceutiques avec succès. Aujourd’hui, les limites de ce système ont été atteintes. Toutes les molécules thérapeutiques ont été trouvées.
Dépasser les limites de la recombinaison
Les chercheurs de l’Institut Pasteur ont donc pensé à une autre approche : celle de la recombinaison. La recombinaison est un mécanisme naturel qui permet le brassage des gènes chez tous les organismes vivants. En biotechnologie, c’est-à-dire provoquée artificiellement (une molécule d’ADN est coupée puis jointe à une autre), la recombinaison se révèle un outil efficace pour générer de nouvelles combinaisons génétiques et favoriser la diversité des molécules produites. Mais elle a ses limites :
- dans les systèmes de recombinaison « séquence-spécifiques », la séquence recombinée ne peut pas être facilement modifiée ou doit rester constante ;
- dans les systèmes de recombinaison dits « homologues », les sites ciblés (des régions de l’ADN) ont une haute similarité.
Ces contraintes structurales réduisent les possibilités d’insérer de l’ADN recombiné dans des régions de l’ADN qui possède déjà une fonction. « Le développement d’un système de recombinaison “site-spécifique”, et non pas “séquence-spécifique”, permettrait d’intégrer des sites de recombinaison dans pratiquement n’importe quelle séquence d’ADN », explique Didier Mazel, responsable de l’unité Plasticité du génome bactérien à l’Institut Pasteur.
Un outil biotechnologique basé sur le système des intégrons
L’équipe de Didier Mazel a concentré ses efforts sur un élément génétique connu pour son rôle dans un mécanisme de recombinaison : les intégrons. « En observant les sites de recombinaison des intégrons, nous nous sommes rendus compte qu’il y avait très peu de spécificité dans les séquences. Seulement 3 ou 4 bases [petites molécules constitutives de l’ADN, NDLR] dont l’éloignement est, de plus, variable », explique Didier Mazel.
Les chercheurs ont pu déterminer les caractéristiques de reconnaissance du système de recombinaison : il se fait au niveau de l’ADN dit « simple brin » et ce n’est pas la structure primaire de l’ADN qui est reconnue mais la secondaire (c’est-à-dire quelques bases qui sortent de la structure en hélice de l’ADN). En décryptant le mode opératoire, il devient possible d’utiliser le pouvoir recombinant des intégrons : « On peut créer artificiellement des combinaisons de gènes qui n’existent pas dans la nature et qui peuvent permettre la synthèse efficace de molécules inédites », poursuit Didier Mazel. La nécessité de produire des nouvelles molécules est aujourd’hui criante face à la crise de l’antibiorésistance.
Le machine-learning à l’appui de la biotechnologie
Dans le cadre de ces travaux, les chercheurs ont dû concevoir un programme informatique qui permet de créer de novo des séquences génétiques ayant un pouvoir de recombinaison. « Au départ, on pensait tout connaître des déterminants de la recombinaison dans ces éléments génétiques sur lesquels nous travaillons depuis plus de 20 ans. En testant nos séquences génétiques, on a constaté que certaines recombinaient bien, d’autres moyennement, et d’autres pas du tout », explique Didier Mazel. En collaboration avec Julia Bernardes du Laboratoire de Biologie Computationnelle et Quantitative, de Sorbonne Universités, son équipe a essayé de comprendre ce qui conditionnait ces variations en appliquant une approche de machine-learning pour extraire des informations en testant différentes hypothèses. « Nous avons découvert de nouveaux déterminants qui améliorent la prédiction d’efficacité de recombinaison des séquences de novo dans les sites », conclut le chercheur.
Source
Structure-specific DNA recombination sites: design, validation and machine learning based refinement, Science Advances, 24 juillet 2020
Aleksandra Nivina1,2,3, Maj Svea Grieb1,2, Céline Loot1,2, David Bikard1,2, Jean Cury1,2,3, Laila Shehata1,2, Juliana Bernardes4 and Didier Mazel1,2
1 Unité Plasticité du Génome Bactérien, Institut Pasteur, 75724 Paris, France
2 CNRS UMR3525, 75724 Paris, France
3 Paris Descartes, Sorbonne Paris Cité, Paris, France
4 Laboratoire de Biologie Computationnelle et Quantitative, Sorbonne Universités, CNRS 19 UMR7238, 75005 Paris, France.
Cette étude entre dans le cadre de l’axe scientifique prioritaire Résistance aux agents antimicrobiens du plan stratégique 2019-2023 de l’Institut Pasteur.