Le 24 janvier 2020, le ministère français chargé de la santé confirmait trois premiers cas de patients touchés par le coronavirus SARS-CoV-2. Le 29 janvier 2020, l’Institut Pasteur, en charge de la surveillance des virus respiratoires en France, a séquencé intégralement le génome du coronavirus dit « 2019-nCoV ». C’est une première en Europe depuis le début de l’épidémie, permise grâce à la Plateforme de microbiologie mutualisée (P2M) de l’Institut Pasteur. P2M est dédiée au séquençage génomique des souches (bactéries, virus, champignons, parasites) que reçoivent les Centres nationaux de référence et les Centres collaborateurs de l’Organisation mondiale de la santé dans le cadre de la surveillance des maladies infectieuses.
Décembre 2019, une épidémie de pneumonies d’allure virale d’étiologie inconnue émerge dans la ville de Wuhan (province de Hubei, Chine).
Le 9 janvier 2020, les autorités sanitaires chinoises et l’Organisation mondiale de la santé (OMS) annoncent la découverte d’un nouveau coronavirus, appelé 2019-nCoV (isolé le 7 janvier), et présenté comme l’agent responsable de ces pneumonies (voir la fiche maladie de l’Institut Pasteur « Coronavirus SARS-CoV-2 »).
Les autorités chinoises partagent, dès le week-end du 11-12 janvier, la séquence complète du génome du coronavirus qu’ils ont détecté dans des échantillons prélevés sur leurs premiers patients. « La séquence du génome des pathogènes est cruciale pour développer des tests de diagnostic spécifiques et identifier les options d’intervention potentielles », souligne Sylvie van der Werf, responsable du Centre national de référence (CNR) virus des infections respiratoires à l’Institut Pasteur.
Vendredi 24 janvier 2020. La détection du virus, confirmée en France.
Vendredi 24, en fin de matinée, des échantillons parviennent à l’Institut Pasteur : trois suspicions de cas possibles (deux patients à Paris, un à Bordeaux). « A partir de ces échantillons prélevés sur ces patients, nous avons détecté le nouveau coronavirus », explique Sylvie Behillil, responsable adjointe du CNR à l’Institut Pasteur.
Dès le vendredi 24 janvier 2020. Le séquençage du génome du virus à l’Institut Pasteur.
Dès le vendredi soir, à partir des échantillons, le séquençage du génome viral est lancé. Le CNR prépare le matériel à séquencer qui sera pris en charge dès le lundi par P2M. Le mardi, en début de soirée, le « run » de séquençage s’achève et l’analyse des données permet de disposer de la séquence du génome complet chez deux des trois premiers cas confirmés en France. « Nous prouvons ainsi l’efficacité du processus d'analyse par séquençage des virus mis en place par le CNR », reprend Vincent Enouf, responsable adjoint du CNR à l’Institut Pasteur.
Mercredi 29 et jeudi 30 janvier 2020. Le séquençage complet est établi puis partagé par l’Institut Pasteur.
La Plateforme de microbiologie mutualisée P2M (voir encadré en bas de page) a aujourd’hui un très haut niveau de performance ; le délai de production des séquences va en moyenne de trois jours (urgences) à dix jours maximum. Ici, en trois jours, la séquence complète est établie (le 29 janvier) : « Nous avons procédé aux analyses de données durant la nuit du mardi au mercredi, corroborées dans la journée du mercredi par des contre-analyses, explique Vincent Enouf. Le séquençage complet a pu être réalisé en trois jours. »
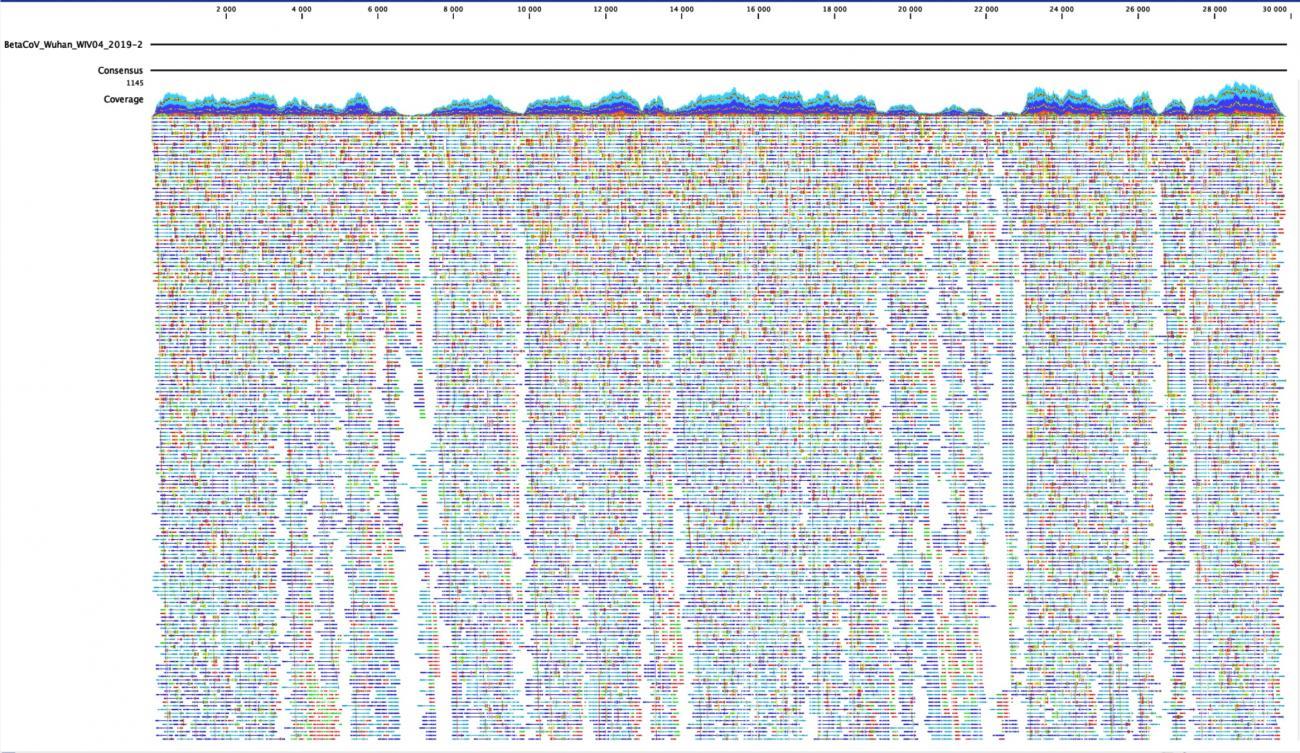

Qu’apprend-on ? « Les séquences sont identiques dans tous nos prélèvements. Un membre du couple a dû contaminer l’autre, c’est le même virus. » Les deux séquences complètes des virus prélevés sur deux des premiers cas français ont été déposées le 30 janvier sur la plateforme du «Global initiative on sharing all influenza data » (GISAID)1, initialement développée pour le partage des séquences et le suivi de l’évolution génétique des virus grippaux, essentiels pour la définition de la composition du vaccin contre la grippe. Un onglet spécial "coronavirus" a été créé pour que la communauté scientifique collabore et avance plus vite.
« Une vingtaine d’autres séquences du génome du nouveau coronavirus ont été établies dans le monde et, en les comparant avec les nôtres, nous nous apercevons qu’elles sont toutes très proches, il n’y a pas beaucoup de diversité dans les virus analysés ce qui va dans le sens que le coronavirus 2019-nCoV n’a pas eu besoin de muter pour s’adapter et se propager », reprend Vincent Enouf.
Le Centre national de référence (CNR) des virus des infections respiratoires, à l’Institut Pasteur (Paris), fait partie des laboratoires référents de l’OMS pour le coronavirus 2019-nCoV.
Au total, huit personnes du CNR et deux de la plateforme de séquençage P2M ont été mobilisées cette semaine, et continuent à l’être pour surveiller l’épidémie en France.
La Plateforme de microbiologie mutualisée (P2M), haute performance, est également ouverte aux CNR externes
Ouverte au séquençage pour des CNR externes, P2M a interagi en 2019 avec 4 CNR non pasteuriens. La plateforme séquence bactéries, virus, parasites et champignons. L’expérience acquise au cours de cinq ans d’activité (depuis 2015) vaut aujourd’hui un très haut niveau de performance, comme l’a montré l’indicateur de taux de réussite au premier « passage » (c’est-à-dire d’obtention d’une séquence de qualité pour une information complète sur le génome) qui s’est élevé à plus de 95 % en 2019. Le délai de production des séquences va de trois jours (urgences) à dix jours maximum.
En 2019, P2M a séquencé environ 25 000 pathogènes. La technique du séquençage génomique augmente le seuil de sensibilité pour la détection des épidémies. En identifiant une épidémie précocement (cas regroupés dans le temps, causés par un même type de pathogène), l’Institut Pasteur permet de mobiliser rapidement les épidémiologistes qui prennent le relais pour en déterminer l’origine, et les autorités qui coordonnent la réponse de santé publique.