La structure des gènes, notamment celle de la séquence de tête de transcription, est très diversifiée chez les champignons. Une nouvelle analyse de données de séquençage par l’unité Biologie des ARN des pathogènes fongiques a révélé que dans les levures pathogènes Cryptococcus, la séquence de tête foisonnait de cadres de lecture ouverts en amont potentiels, qui régulent à la fois l’expression des gènes et la diversité des protéines.
Les espèces pathogènes de Cryptococcus sont responsables de près de 200 000 décès chaque année dans le monde. Une meilleure connaissance de la structure des gènes permettrait de comprendre leur régulation et leur impact individuel sur la virulence. Dans cette étude, les chercheurs de l’unité Biologie des ARN des pathogènes fongiques ont exploité une multitude de données de séquençage d’ARN variées afin de réannoter précisément les extrémités de chaque gène codant chez deux champignons pathogènes (à savoir Cryptococcus neoformans et C. deneoformans). Étonnamment, l’analyse de la structure des séquences de tête de transcription de Cryptococcus a révélé la présence de plus de 10 000 cadres de lecture ouverts en amont (uORF) potentiels qui, chez ces champignons, démontrent une contribution majeure à la répression translationnelle.
La synthèse des protéines eucaryotes débute au niveau d’un codon d’initiation défini par un AUG et le contexte de la séquence Kozak qui l’entoure, mais des recherches sur S. cerevisiae suggèrent que ce contexte n’a que peu d’importance chez les champignons. Néanmoins, chez Cryptococcus, l’utilisation des uORF dépend du contexte de la séquence Kozak de son codon d’initiation, et les uORF aux contextes forts favorisent la dégradation des ARNm non-sens. L’exploitation alternative du site de démarrage de la traduction pourrait également se révéler un moyen de diversifier les protéomes de ces levures. Ainsi, beaucoup d’ARNm de Cryptococcus codent des protéines à double localisation prédite, dont de nombreuses aminoacyl-ARNt synthétases, dans lesquelles un codon d’initiation AUG partiellement fonctionnel est suivi d’un AUG encadré à fort contexte Kozak, séparé par une séquence ciblant les mitochondries (Figure). En revanche, chez Saccharomyces cerevisiae, les séquences de tête de transcription sont courtes et comptent moins de 1 000 uORF. L’analyse d’autres espèces fongiques indique que cette double localisation pourrait également se trouver communément dans la moisissure particulière Neurospora crassa. Une corrélation est établie entre la régulation contrôlée par Kozak et les insertions de facteurs d’initiation de la traduction dans des régions déterminant la fidélité en contact avec l’ARNt initiateur. Ainsi, chez les champignons, le contexte du codon d’initiation est un signal qui programme l’expression et les structures des protéines.
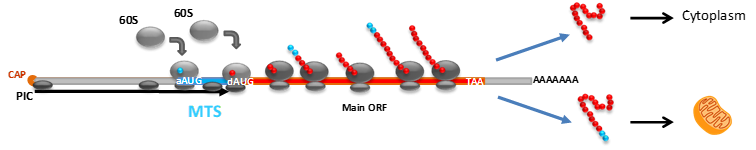
L’utilisation alternative du codon d’initiation selon le contexte de la séquence et de la position peut alors réguler le ciblage de leurs protéines
Nucleic Acids Res. 2020 Mar 18; 48(5): 2312–2331. doi: 10.1093/nar/gkaa060